【佳学基因检测】结直肠癌肿瘤基因检测的方法与科学流程
结直肠癌(CRC)是全球范围内最常见的癌症之一,其发生和发展涉及多种遗传、环境及生活方式因素。近年来,随着肿瘤基因组学的进展,基因检测技术成为早期诊断、预后评估及个性化治疗的重要手段。结直肠癌的肿瘤基因检测通过识别与肿瘤发生、进展和药物反应相关的基因突变和变异,提供了对肿瘤生物学的深刻理解。这些检测方法主要包括基因突变分析、扩增片段长度多态性(PCR)、下一代测序(NGS)、全基因组关联研究(GWAS)等。通过对肿瘤组织和血液样本的检测,科研人员能够发现与结直肠癌相关的特定基因变异,如APC、KRAS、BRAF等突变,这些信息为疾病的早期诊断、风险评估以及选择个性化治疗方案提供了重要依据。同时,基因检测还能帮助预测患者对化疗、靶向治疗等治疗方式的反应,有助于提高治疗效果和患者的生存质量。因此,肿瘤基因检测在结直肠癌的临床应用中具有重要价值,推动了精准医学的发展。
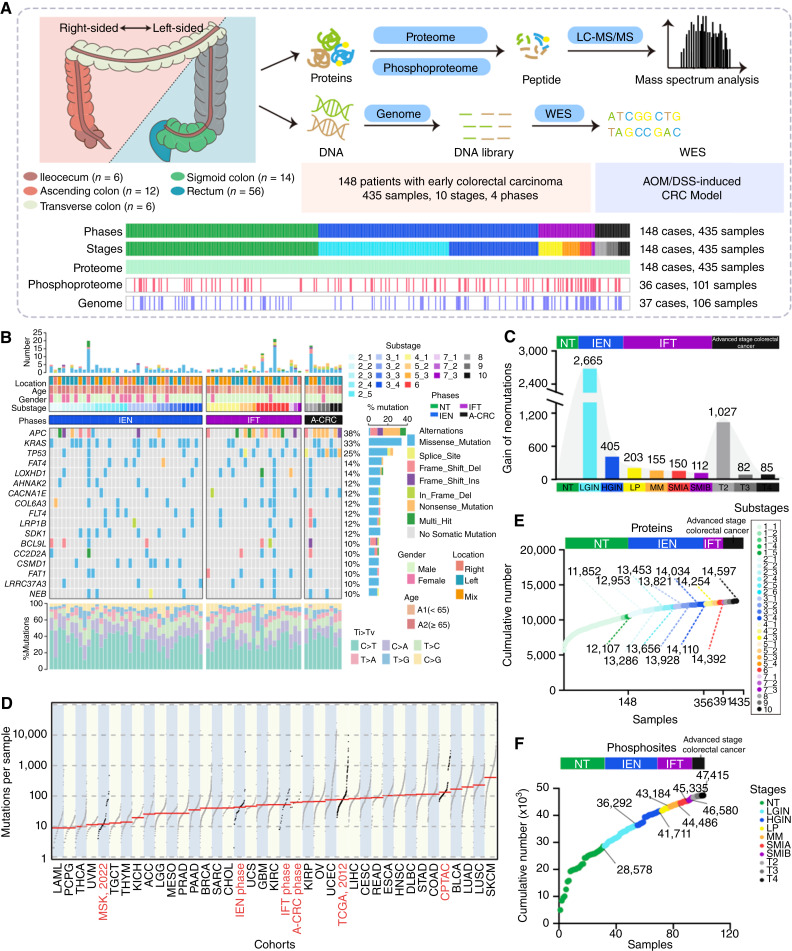
早期结直肠癌队列的构建
结直肠癌肿瘤基因检测连续纳入了 2018 年 1 月至 2018 年 12 月在复旦大学附属中山医院接受内镜黏膜下剥离术治疗的 600 例疑似结直肠病变的结直肠癌患者。正如结直肠癌的靶向用药基因解码基因检测的的方法介绍中的描述,在患者选择上没有偏倚,且所有患者均未接受过放疗或化疗等先前治疗。118 例早期结直肠癌病例符合纳入标准。482 例排除患者中,107 例诊断为非肿瘤(NT)病变,65 例患有间质瘤,110 例患者因无法获得正常组织样本而被排除,200 例样本未通过病理质量检查(如肿瘤细胞比例 < 80%)。随后,对 30 例未接受新辅助治疗且接受手术切除的晚期结直肠癌病例进行筛查。因此,结直肠癌靶向药物基因解码基因检测共选择了 148 名结直肠癌患者构建结直肠癌队列。所有病例均根据美国癌症联合委员会第八版肿瘤-淋巴结-转移分期系统进行分期。结直肠癌肿瘤靶向用药基因检测符合赫尔辛基宣言 II 的伦理标准,并经复旦大学中山医院机构审查委员会批准。所有患者在进行任何特定研究调查之前均提供了书面知情同意书。所有患者在进行任何特定研究调查之前均提供了知情同意书。
结直肠癌肿瘤靶向药物基因检测的所有样本均符合以下标准:首先,根据世界卫生组织的分类,所有样本均保存完好,并由两名以上的胃肠道病理专家进行系统评估,以确认组织病理学诊断和任何变异组织学,他们根据肿瘤含量(> 95%)、肿瘤坏死的存在和程度(< 5%)以及侵入固有肌层的迹象确定可接受的组织片段。其次,应用以下标准:(i)成功提取 DNA 和蛋白质;(ii)正常组织中没有肿瘤细胞。
根据世界卫生组织和日本的病理诊断标准,早期结直肠癌队列中的所有亚期均纳入四个 TNM 分期:T0(正常上皮,n = 166)、T1(T1a/b 癌症,n = 239)、T2(n = 10)、T3(n = 10)和 T4(n = 10)。T1 被细分为低级别 IEN [LGIN;2_1(n = 37)、2_2(n = 29)、2_3(n = 20)、2_4(n = 22)、2_5(n = 4)和 2_6(n = 1)]、高级别 IEN [HGIN; 3_1 ( n = 20)、3_2 ( n = 21)、3_3 ( n = 20) 和 3_4 ( n = 16)]、LP 期 [4_1 ( n = 2)、4_2 ( n = 18) 和 4_3 ( n = 1)]、MM 期 [5_1 ( n = 1)、5_2 ( n = 11)、5_3 ( n = 1) 和 5_4 ( n = 1)],以及粘膜下浸润癌期,即 SMIA [6 ( n = 11)] 和 SMIB 期 [7_1 ( n = 1)、7_2 ( n = 1) 和 7_3 ( n = 1)]。所有样本分为四个阶段:NT期、IEN期(包括LGIN和HGIN期)、IFT期(从LP至SMIB期)、以及晚期结直肠癌期(从T2至T4期)。
福尔马林固定、石蜡包埋标本的采集和处理
所有福尔马林固定石蜡包埋 (FFPE) 标本均由复旦大学中山医院制备和提供。如肿瘤的靶向用药基因检测基因检测所述,对于临床样本制备,对 FFPE 块的载玻片 (10 μm 厚) 进行大体解剖,用二甲苯脱蜡,并用乙醇清洗。将 FFPE 块切成 3 μm 厚的切片,进行苏木精和伊红染色。所有亚期标本均由两名以上经验丰富且获得委员会认证的胃肠道病理学家刮取、评估和确认,并将材料分装并储存在 –80°C 下,直至进一步处理。
左侧结肠直肠癌和右侧结肠直肠癌样本
根据结直肠癌患者病变部位,将其分为三组:单独左侧结直肠癌组,仅患有左侧结直肠癌;单独右侧结直肠癌组,仅患有右侧结直肠癌;混合组,即左侧和右侧结直肠癌均有记载。在主要队列中,混合组记录了左侧和右侧结直肠癌。结直肠癌肿瘤基因检测有效性仅收集其中一个部位(左侧或右侧)的病变,收集肿瘤位置相应侧的正常组织。在验证队列中,混合组同时患有左侧和右侧结直肠癌,收集肿瘤组织和相应的正常组织。验证队列中所有结直肠癌样本(n = 60)均切成 3 mm 厚的切片,然后在苏木精和伊红染色的切片上进行标记。所有正常/肿瘤样本均从 FFPE 载玻片上分别解剖,并由两名以上经验丰富的胃肠病理学家进行评估。
全外显子组测序
全外显子组测序(WES)由佳学基因医学技术(北京)有限公司完成。如肿瘤基因解码基因检测中所述,从FFPE肿瘤组织样本中采集DNA,从NT组织样本中获得匹配的种系DNA。对37例106个样本进行了WES分析,方法的细节由佳学基因医学技术(北京)有限公司提供。使用Qubit 2.0(Thermo Fisher Scientific)对得到的序列文库(双端序列和两端之间的插入DNA)进行定量,使用Agilent 2100生物分析仪(RRID:SCR_018043)测定插入片段大小。使用碱基调用从原始图像数据中获取原始数据(测序读取)。
DNA 提取和 DNA 鉴定
对37例结直肠癌病例的106个样本进行了WES分析,方法学细节由佳学基因靶向药物基因检测提供。所有样本首先用二甲苯脱蜡,然后在1%琼脂糖凝胶上监测DNA降解和污染。随后,使用Qubit DNA分析在Qubit 2.0荧光计(Invitrogen)中测量DNA浓度。每个样本至少使用0.6 μg基因组DNA作为DNA样本制备的输入。
文库制备
方法学细节由佳学基因医学技术(北京)有限公司提供。每个样本的基因组 DNA 量为 0.6 μg,用于 DNA 样本制备。按照制造商的建议,使用 Agilent SureSelect Human All Exon 试剂盒(安捷伦科技)生成测序文库,并为每个样本添加索引代码。
使用流体力学剪切系统(Covaris)进行片段化,随机生成180至280 bp的片段。剩余的突出端通过外切酶/聚合酶活性转化为平端。在DNA片段的3′端腺苷酸化后,连接接头寡核苷酸。在PCR中选择性富集两端连接有接头分子的DNA片段。PCR后,用生物素标记的探针将文库与液相杂交,然后使用含有链霉素的磁珠捕获基因的外显子。在PCR中富集捕获的文库以添加索引标签以准备测序。使用AMPure XP系统(Beckman Coulter)纯化产物,并使用Agilent Bioanalyzer 2100系统上的Agilent高灵敏度DNA分析进行定量。
使用 HiSeq PE 聚类试剂盒(Illumina),按照制造商的说明,在 cBot 聚类生成系统上对索引编码样本进行聚类。聚类生成后,在 Illumina HiSeq 平台上对 DNA 文库进行测序,并生成 150 bp 的双端读取。
数据处理和分析的质量控制
具体实验与操作方法由佳学基因医学技术(北京)有限公司提供。双端测序(PE150)在 Illumina HiSeq(Illumina NovaSeq 6000)上进行。使用 Qubit 2.0(Thermo Fisher Scientific)对得到的序列文库(双端序列和两端之间的插入片段 DNA)进行定量,使用 Agilent 2100 生物分析仪测定插入片段大小。从 HiSeq 平台获取的原始荧光图像文件通过碱基调用转换为短读(原始数据),并将这些短读记录为 FASTQ 格式,其中包含序列信息和相应的测序质量信息。使用碱基调用从原始图像数据中获取原始数据(测序读段)。
质量控制:
- (一)如果任一读取包含适配器污染(与适配器对齐的核苷酸 > 10 个,允许≤10%的错配),则丢弃配对读取;
- (二)如果任一读取中超过 10% 的碱基不确定,则丢弃配对读取;
- (三)如果任一读取中低质量(Phred 质量 < 5)碱基的比例超过 50%,则丢弃配对读取。
所有下游生物信息学分析均以高质量的干净数据为基础,保留干净数据,同时计算并汇总质控统计数据,包括总read数、原始数据、原始深度、测序错误率、Q30(Phred-scaled质量得分>30的碱基百分比)reads百分比、QC内容分布等。
读取映射到参考序列
方法的细节由佳学基因医学技术(北京)有限公司提供。使用 Burrows–Wheeler Aligner(BWA)软件(RRID:SCR_010910)将有效测序数据比对到参考人类基因组(UCSC hg19),以获得以 BAM 格式存储的原始比对结果。如果一个或一对读取被映射到多个位置,BWA 采用的策略是选择最可能的位置。如果存在两个或两个以上最可能的位置,BWA 会随机挑选一个,然后使用 SAMtools(RRID:SCR_002105)和 Picard(http://broadinstitute.github.io/picard/,RRID:SCR_006525)对 BAM 文件进行排序,并进行重复标记、局部重新比对和碱基质量重新校准,以生成最终的 BAM 文件,用于计算序列覆盖率和深度。由于存在错配(包括真突变和测序错误)以及 PCR 扩增导致的重复,映射步骤非常困难。这些重复读取没有信息量,不应被视为变异的证据。使用 Picard(RRID:SCR_006525)标记这些重复,以便进行后续分析。
体细胞突变的检测和调用
方法学细节由佳学基因医学技术(北京)有限公司提供。使用BWA和Samblaster进行基因组比对,使用MuTect软件(RRID:SCR_000559)定位体细胞单核苷酸变异位点,使用Strelka检测体细胞INDEL信息。本文使用的统计数据包括适度t统计量和Fisher精确检验。
新突变的获得
为了探究结直肠癌致癌过程中各个阶段的突变情况,首先统计每个阶段的突变,然后重点关注新突变的获得。在结直肠癌基因解码基因检测中,新突变意味着只在某个阶段发生,而不是在更早的阶段存在。例如,如果AFP在LGIN阶段未发生突变,而在HGIN阶段发生突变,则AFP在HGIN阶段即为新突变。某一阶段的新突变也可以反映特定突变在结直肠癌进展中的作用。
检测到的突变对蛋白质和磷蛋白水平的影响
体细胞拷贝数改变及其对蛋白质组的影响
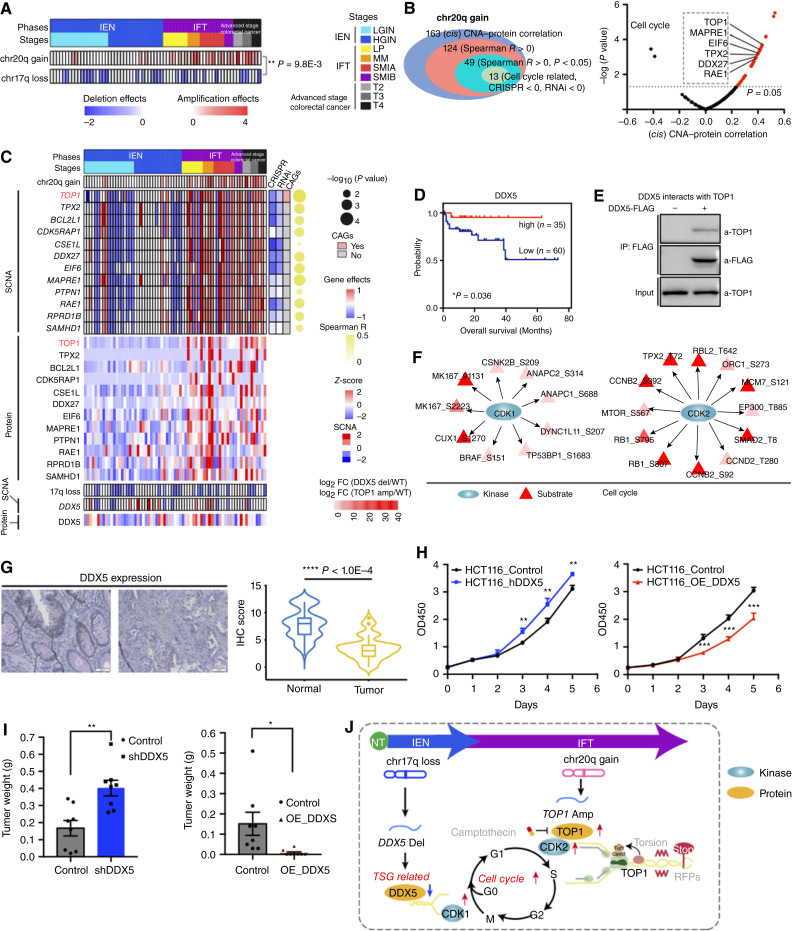
对于体细胞拷贝数变异 (SCNA) 分析,使用了在体细胞突变检测流程中处理的 WES 衍生 BAM 文件。为了研究SCNA 在 chr20q 增加和 chr17q 丢失时的顺式/反式效应,重点研究了在 SCNA 和蛋白质水平上均检测到的基因,然后计算了斯皮尔曼相关系数 (FDR < 0.05)。
定义癌症相关基因
癌症相关基因 (CAG) 是根据 Bailey 及其同事定义并由 Mertins 及其同事列出的基因汇编而成的。CAG 列表请查阅《人体基因序列变与人的疾病表征》数据库。
蛋白质提取和胰蛋白酶消化
如前文所述,所有标本均采用显微切割技术进行切割,收集于1.5 mL EP管中,保存于-80℃冰箱中。每片FFPE切片厚度为10 μM,每个亚组标本细胞数不超过10,000个。
将 50 μL Tris(2-羧基乙基)-膦-HCl 缓冲液(2% 脱氧胆酸钠盐(Solarbio,目录号 D8330)、40 mmol/L 2-氯乙酰胺(Aldrich,目录号 22790-250G-F)、100 mmol/L Tris-膦盐酸盐(Amresco,目录号 0497)、10 mmol/L(2-羧基)-膦盐酸盐(Aldrich,目录号 4706-10G)和 1 mmol/L 苯甲基磺酰氟(Amresco,目录号 M145-5G)与质谱 (MS) 级水(JT Baker,目录号 4218-03,pH 8.8)混合,加入装有制备样品的 1.5 mL EP 管中,然后在99°C 金属浴中加热 30 分钟。冷却至室温后,向每管中加入 3 μg 胰蛋白酶(Promega,目录号 V528A),并在 37°C 培养箱中消化 18 小时。然后,向每管中加入 13 μL 10% 甲酸 (FA;Sigma,目录号 F0507),涡旋 3 分钟,然后离心 5 分钟(12,000 g)。之后,使用新的 1.5 mL 管和 350 μL 缓冲液[0.1% FA 在 50% 乙腈 (ACN;JT Baker,目录号 9830-03)] 收集上清液进行提取(涡旋 3 分钟,然后在 12,000 g下离心5 分钟)。将上清液转移到新管中并在 60°C 下真空干燥。干燥后用100 μL 0.1% FA溶解多肽,旋涡混合,离心3 min(12 000 g),将上清液收集至新管中脱盐。脱盐前需用两片3M C18 盘片对柱子进行活化,活化液为:90 μL 100% ACN 2次,90 μL 50%和80% ACN 依次活化1次,90 μL 50% ACN 1次。然后将上清液装入柱子2次,再用90 μL 0.1% FA 净化2次。最后加入90 μL 洗脱缓冲液(0.1% FA in 50% ACN)洗脱2次,仅收集流出液进行MS分析。将收集液在60°C下真空干燥(约1.5小时)。
使用 LC-MS/MS 分析进行蛋白质组分析
如我们之前的研究(16、17、21)中所述,对于样品的蛋白质组学分析,使用 Q Exactive HF-X 混合四极杆轨道阱质谱仪(赛默飞世尔科技)结合高效液相色谱系统(EASY-nLC 1200,赛默飞世尔科技)对肽进行分析。将干燥的肽样品重新溶解在溶剂 A(水中 0.1% FA)中,然后使用溶剂 A 将其装入 2 cm 自填充捕获柱(内径 100 μm,实践尺寸 3 μm ReproSil-Pur C18-AQ 珠,自制,SunChrom),并在 150 μm 内径、长度 15 cm 的柱上进行分离(实践尺寸 1.9 μm ReproSil-Pur C18-AQ 珠,自制,SunChrom),梯度洗脱时间为 150 分钟(溶剂 A:水中 0.1% FA;溶剂 B:80% ACN 中的 0.1% FA),恒定流速为 600 nL/分钟(0-150 分钟,0 分钟,4% B;0-10 分钟,4%-15% B;10-125 分钟, 15%–30% B;125–140分钟,30%–50% B;140–141分钟,50%–100% B;141–150分钟,100% B)。将洗脱的肽在2.0 kV下电离并引入质谱仪)。MS在数据依赖性采集模式下执行。对于MS1光谱全扫描,使用Orbitrap质谱分析仪以120,000的高分辨率采集m/z范围为300至1,400的离子。自动增益控制(AGC)目标值设置为3E6。最大离子注入时间为80 ms。MS2光谱采集在离子阱模式下快速进行。选择前体离子并以更高能量碰撞解离碎裂,标准化碰撞能量为27%。使用离子阱质谱分析仪分析碎片离子,AGC 目标为 5E4。MS2 的最大离子注入时间为 20 毫秒。触发 MS/MS 扫描的肽在 12 秒内被动态排除在进一步的 MS/MS 扫描之外。
对于磷酸化蛋白质组学样品,使用 Q Exactive HF-X 混合四极杆轨道阱质谱仪(赛默飞世尔科技)结合高效液相色谱系统(EASY-nLC 1200,赛默飞世尔科技)对肽进行分析。将干燥的肽样品重新溶解在溶剂 A(水中含 0.1% FA)中,使用溶剂 A 将其装入 2 cm 自填充捕获柱(内径 100 μm,实践尺寸 3 μm ReproSil-Pur C18-AQ 珠,自制,SunChrom),并在 150 μm 内径、长度 30 cm 的柱上进行分离(实践尺寸 1.9 μm ReproSil-Pur C18-AQ 珠,自制,SunChrom),在 150 分钟的梯度(缓冲液 A:水中含 0.1% 甲酸;缓冲液 B:80% ACN 中含 0.1% FA)下以 600 nL/分钟的恒定流速进行分离(0-150 分钟,0 分钟,4% B;0-10 分钟,4%-15% B;10-125 分钟, 15%–30% B;125–140 分钟,30%–50% B;140–141 分钟,50%–100% B;141–150 分钟,100% B)。洗脱的磷酸肽被电离并使用 Q Exactive HF-X 混合四极杆轨道阱质谱检测。质谱采集范围为 m/z 300 至 1,400,分辨率为 120,000(AUG 目标值为 3E+06,最大注射时间为 80 毫秒)。对于 MS2 扫描,在标准化碰撞能量为 30% 的情况下执行更高能量碰撞解离碎片。MS2 AGC 目标设置为 5E4,最大注射时间为 100 毫秒。选择肽模式进行单同位素前体扫描,并启用电荷状态筛选以拒绝未分配的 1+、7+、8+ 和 >8+ 离子,动态排除时间为 40 秒,以区分±10 ppm 之间先前分析的离子。
磷酸肽富集与分析
所有符合要求的分析数据均在 Firmiana 平台上根据 NCBI 中的人类 RefSeq 蛋白质数据库(更新于 2013-07-04;RRID:SCR_003496)进行处理。由于样本量有限,只有来自 36 名结肠直肠癌患者的 101 个样本适合进行磷酸化蛋白质组分析:NT 期(n = 31)、LGIN 期(n = 23)、HGIN 期(n = 17)、LP 期(n = 6)、MM 期(n = 7)、SMIA 期(n = 6)、SMIB 期(n = 3)、T2 期(n = 3)、T3 期(n = 3)和 T4 期(n = 2;补充表 S1D)。
如肿瘤发生的基因解码技术方案中所述,根据制造商的说明,使用 Fe-NTA 磷酸肽富集试剂盒(Thermo Fisher Scientific,目录号 A32992)制备磷酸化蛋白质组样品。简而言之,将 2 mg 肽悬浮在 200 μL 结合/洗涤缓冲液中,并加载到平衡的旋转柱中。轻轻拍打使树脂与样品混合。将混合物孵育 30 分钟,以 1,000 × g离心 30 秒以弃去流出物。然后用 200 μL 结合/洗涤缓冲液洗涤柱子,以 1,000 × g离心30 秒,共 3 次,再用 200 μL LC-MS 级水洗涤一次。用100 μL洗脱缓冲液洗脱磷酸肽,以1,000 × g离心30秒,重复2次。将磷酸肽干燥后进行LC-MS/MS分析。
整体蛋白质组数据和磷酸化蛋白质组数据的量化
如结直肠癌的致病基因鉴定基因解码所述,所有 MS 原始文件均在 Firmiana 平台(一站式蛋白质组学云平台:http://www.firmiana.org )上进行处理,并在 Mascot 搜索引擎(版本 2.3,Matrix Science Inc.,RRID:SCR_014322)中根据 NCBI 人类 RefSeq 蛋白质数据库(2013 年 4 月 7 日更新,32,015 个条目)进行搜索。使用胰蛋白酶作为蛋白水解酶,最多允许两次漏切。脲甲基 (C) 被视为固定修饰。对于蛋白质组分析数据,可变修饰是氧化 (M) 和乙酰化(蛋白质 N 端)。对于磷酸化蛋白质组数据,可变修饰是氧化 (M)、乙酰化(蛋白质 N 端)和磷酸化 (S/T/Y)。所有已鉴定的肽均在 Firmiana 平台上定量,峰面积由其 MS1 强度得出。通过 Q-Exactive HFX 收集的母体和产物的质量公差分别为 20 ppm 和 50 mmu。母体离子分电荷限制为 +2、+3 和 +4。肽谱匹配和蛋白质的 FDR 设定为最大 1%。我们的研究采用了无标记蛋白质定量,即所谓的 iBAQ 算法,该算法将蛋白质丰度(由已鉴定肽的强度得出)除以理论上可观察到的肽的数量。然后,总量的分数(定义为蛋白质的 iBAQ 除以一个样本中所有已鉴定蛋白质的总 iBAQ)用于表示特定蛋白质在样本中的标准化丰度。
数据归纳
如结直肠癌的致病基因鉴定基因解码所述,对于研究中的缺失值,首先应用了运行间匹配算法,该算法已被证明是一种有效的填充缺失值的技术。简而言之,根据样品中的常见识别肽建立了一个动态回归函数。根据相关值R 2,函数选择线性或二次函数进行回归计算相应隐藏肽的保留时间(RT),并根据 m/z 和计算出的 RT 检查提取离子色谱图的存在。该函数评估所显示的提取离子色谱图的峰面积值。这些峰面积值被视为相应蛋白质的一部分。
层次聚类分析
如结直肠癌的致病基因鉴定基因解码所述所述,在 R (版本 3.5.1) 中实现了层次聚类分析和主成分分析 (PCA),以评估我们的蛋白质组数据集中关于以下两个变量的批次效应:批次身份和样本类型 (亚阶段/亚型/面板)。对于层次聚类分析,首先研究了同一亚阶段中样本的成对 Spearman 相关系数。为此,同一类型的样本表现出较高的相似性,而不同亚型的样本明显不同。此外,使用了平均链接算法,以一减去 Spearman 相关系数作为相异度度量。
在全局热图中,全局蛋白质组表达矩阵中的每个蛋白质表达值都转换为所有样本的Z分数。对于样本和蛋白质聚类,距离设置为“欧几里得”距离,权重方法为“完整”。使用 R 包 pheatmap(版本 1.0.12,RRID:SCR_016418)对Z分数转换后的矩阵进行聚类。
差异蛋白质组学分析和通路富集
为了比较结直肠癌进展过程中不同阶段的差异表达蛋白(DEP)(图1),关注每个阶段的蛋白质丰度(平均值),这些蛋白质由京都基因和基因组百科全书(KEGG;RRID:SCR_012773)/基因本体(GO;RRID:SCR_002811)数据库和 ConsensusPathDB(http://cpdb.molgen.mpg.de/,RRID:SCR_002231 )富集。然后,我们注释了信号通路(调整。FDR < 0.05)并手动检查通路相关蛋白,然后估计这些蛋白是否与结直肠癌分期显着相关(Kruskal-Wallis 检验,调整。P < 0.05)。
图 1.结直肠癌进展中的多组学概况。
为了分析KRAS和BRAF突变的不同功能(图2),分别对KRAS突变组与野生型(WT)组、BRAF突变组与WT组进行DEP检验(Wilcoxon秩和检验,FDR < 0.05,Mut vs. WT比例≥2),然后基于基因集富集分析(GSEA)进行比较分析。
图 2.KRAS和BRAF突变对结直肠癌进展的影响。
为了在磷蛋白水平分析KRAS和BRAF突变如何在结直肠癌进展中调控不同的功能(图2 ),我们分别在KRAS突变组与WT组之间、BRAF突变组与WT组之间进行了DEP分析(Wilcoxon秩和检验,FDR < 0.05,Mut vs. WT比例≥ 2,Phos vs. Pro≥ 2),并对KRAS突变组和BRAF突变组的磷酸化蛋白质组进行激酶-底物富集分析(KSEA),建立KRAS突变-激酶-底物网络和BRAF突变-激酶-底物网络。
为了在磷酸化蛋白水平分析DDX5缺失和TOP1扩增对细胞周期的影响(图3),对DDX5缺失组与WT组、TOP1扩增组与WT组进行DEP检验(Wilcoxon秩和检验,FDR < 0.05,Del/Amp vs. WT比例≥2,Phos vs. Pro≥2),然后应用KEGG/GO数据库进行通路富集,并对DDX5缺失组和TOP1扩增组的磷酸化蛋白质组进行KSEA,建立DDX5缺失-激酶-底物网络和TOP1扩增-激酶-底物网络。
图 3.
chr17q 丢失和 chr20q 增加在结直肠癌进展中的作用。A ,热图显示 chr20q 增加和 chr17q 丢失在结直肠癌进展中的关联(Fisher 精确检验)。B ,维恩图(左)和火山图(右)描绘了通过 chr20q 增加(Spearman 相关性)将分析限制在多种数据类型中的显着顺式效应。C ,热图显示显着相关基因在 chr20q 增加和 chr17q 丢失时对其相应蛋白质表达的顺式 SCNA 蛋白质调控(Spearman 相关性)。D ,DDX5与结直肠癌患者的预后呈正相关(对数秩检验)。E ,共免疫沉淀结果显示 HEK293T 细胞中的 DDX5 和 TOP1 相互作用。F ,细胞周期中的 CDK1(左)/CDK2(右)底物相互作用网络。G , IHC 染色(左)和箱线图(右)显示 DDX5 的表达(t检验,n = 244)。H和I, DDX5 敲低(左)和过表达(右)对细胞增殖(t检验;H)和肿瘤重量(n = 32;I)的影响。J , chr17q 丢失和 chr20q 增加在结直肠癌进展中的作用的简要总结。误差线,平均值 ± SEM。*,P < 0.05;**,P < 1.0E−2;***,P < 1.0E−3;****,P < 1.0E−4;ns.,不显著,> 0.05。OD,光密度。
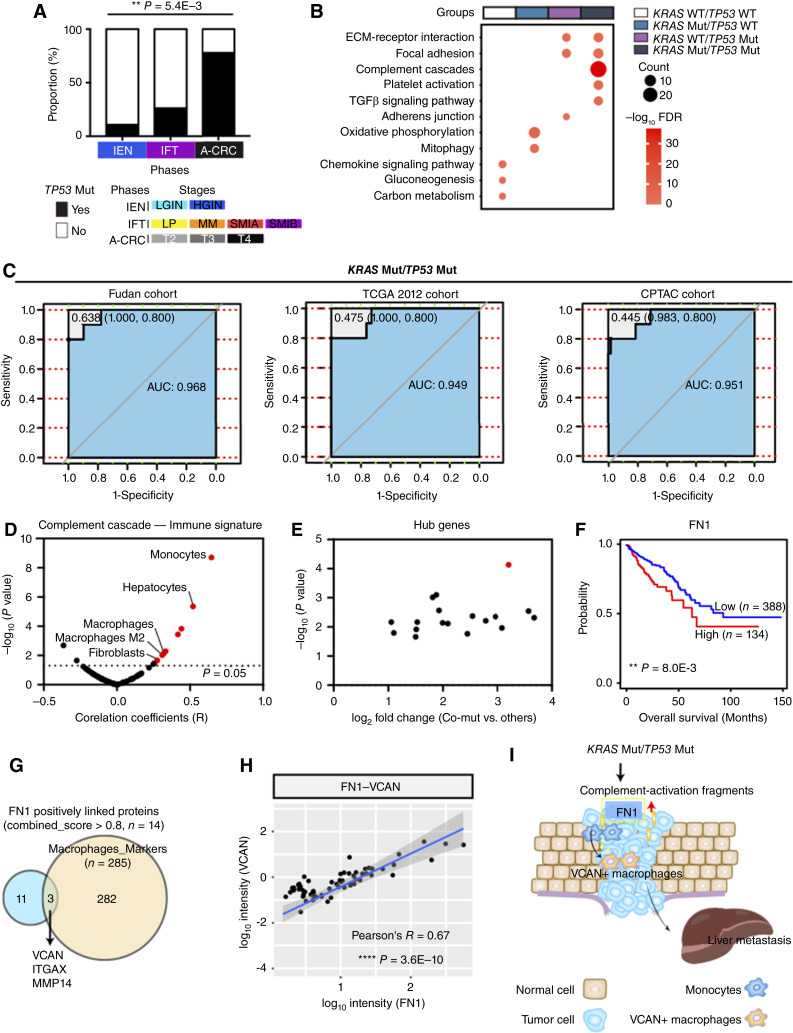
为分析KRAS与TP53共突变的功能(图4),建立4组:KRAS WT和TP53 WT(KRAS WT/ TP53 WT)组、KRAS WT和TP53 Mut(KRAS WT/ TP53 Mut)组、KRAS Mut和TP53 WT(KRAS Mut/ TP53 WT)组、KRAS Mut和TP53 Mut(KRAS Mut/ TP53 Mut)组。利用4组间的DEPs进行通路富集分析(Kruskal–Wallis检验,FDR < 0.05),然后应用KEGG/GO数据库进行通路富集分析。
图4:TP53突变对结直肠癌进展的影响。A ,结直肠癌各期TP53突变比例(Fisher 精确检验)。B ,四组中的主要通路。C ,复旦队列和其他结直肠癌队列中 20 种蛋白质的 ROC 曲线。D ,火山图显示补体级联与结直肠癌进展中免疫特征之间的(Spearman)相关性。E ,火山图显示KRAS和TP53共突变组与其他组之间这 20 种蛋白质的log 2倍变化和P值(Wilcoxon 秩和检验)。F ,结直肠癌患者中 FN1 的生存分析(对数秩检验)。G ,维恩图绘制了 FN1 正相关蛋白与巨噬细胞标志物之间的覆盖率。H,散点图显示复旦队列中log 10 FN1 和 log 10 VCAN 在蛋白质水平上的(Pearson) 相关性。I ,简要总结 FN1 在结直肠癌肝转移中的作用。*,P < 0.05;**, P < 1.0E-2;***, P < 1.0E-3;****, P < 1.0E-4;ns,无显著性,> 0.05。
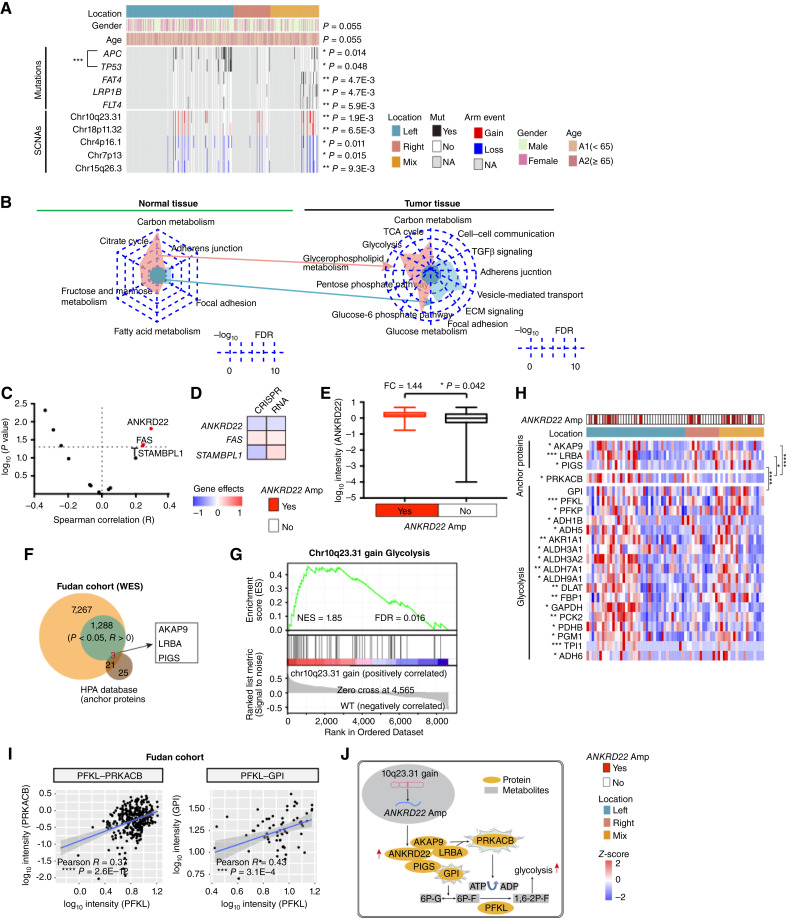
为了分析左侧结直肠癌单独组和右侧结直肠癌单独组之间的差异(图5),收集了435个样本的主要队列和另一个包含60个样本的独立验证队列,并在左侧结直肠癌单独组和右侧结直肠癌单独组之间应用DEP(Wilcoxon秩和检验,FDR < 0.05,左侧结直肠癌与右侧结直肠癌的比例≥2或≤0.5)。在混合组中,在正常组织和肿瘤组织之间应用DEP(Wilcoxon秩和检验,FDR < 0.05,正常与肿瘤的比例≥2或≤0.5)。为了分析左侧结直肠癌/右侧结直肠癌与混合组的差异,采用左侧结直肠癌/右侧结直肠癌与混合组之间的DEP(Wilcoxon秩和检验,FDR < 0.05,左侧结直肠癌/右侧结直肠癌与混合比≥2或≤0.5),然后应用KEGG/GO数据库进行通路富集。
图 5.
基于位置的结肠直肠癌的多种特征。A 、基于位置的结肠直肠癌中显着相关的临床特征、突变和 SCNA(Fisher 精确检验)。B 、左侧结肠直肠癌和右侧结肠直肠癌的正常(左)和肿瘤(右)组织中的特定通路。C 、火山图显示SCNA 的顺式相关性以及 chr10q23.31 处其基因的相关 −log 10(P值)(Spearman 相关性)。D 、依赖图支持的面板显示在通过 RNAi 或 CRISPR 消耗指定基因后所有可用结肠直肠癌细胞系的平均相对生存率。E 、ANKRD22扩增对其对应蛋白表达的影响(Wilcoxon 秩和检验)。F 、维恩图描绘了与 ANKRD22 显着相关并被纳入 HPA 锚蛋白数据库的蛋白质(Pearson 相关性)。G,与 WT 比较,chr10q23.31 增益组糖酵解的 GSEA 图(KEGG 基因集)。H ,热图显示ANKRD22扩增组中糖酵解过度表达。I ,散点图显示蛋白质水平上 log 10 PFKL 和 log 10 PRKACB/GPI 表达之间的 (Pearson) 相关性。J ,简要总结了 chr10q23.31 增益对结直肠癌进展的影响。误差线,平均值 ± SEM。*,P < 0.05;**, P < 1.0E−2;***, P < 1.0E−3;****, P < 1.0E−4;ns,不显著,> 0.05。
为了分析基于 CMS 和 CRIS 分类的进展路径(图 6),我们评估了结直肠癌进展过程中不同阶段的 DEP(Kruskal-Wallis 检验,FDR < 0.05),然后通过 KEGG/GO 数据库和 ConsensusPathDB(http://cpdb.molgen.mpg.de/)进行富集。然后我们注释了信号通路(FDR < 0.05)并手动检查了通路相关蛋白,然后估计这些是否与结直肠癌的分期显着相关(Kruskal-Wallis 检验,FDR < 0.05)。
图 6.
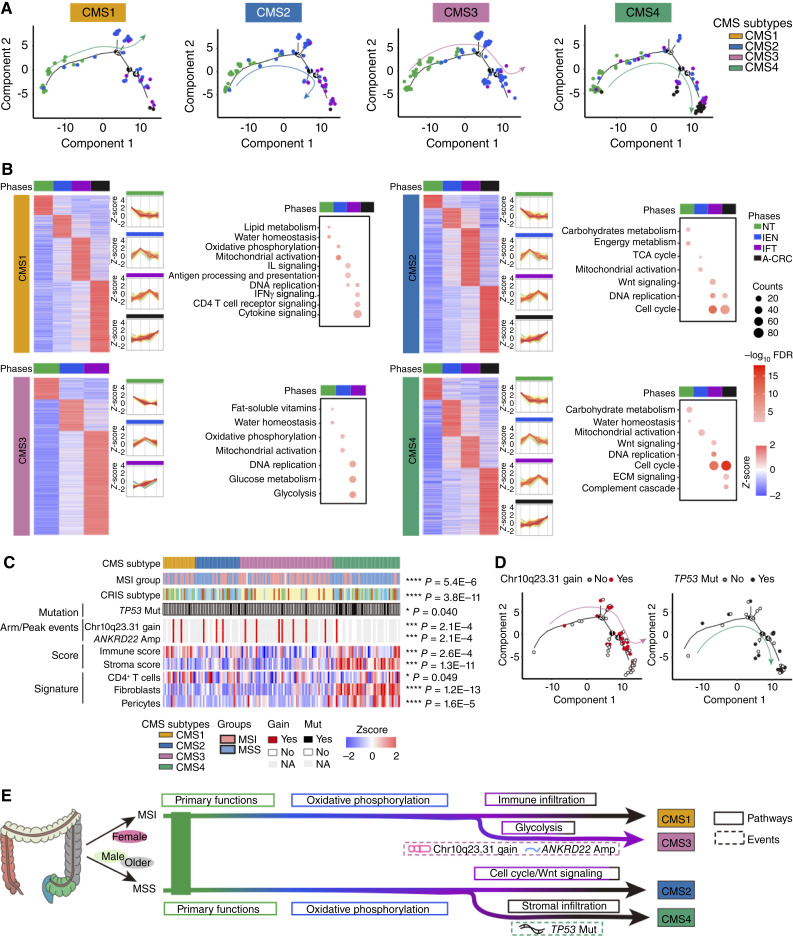
基于 CMS 分类的结肠直肠癌致癌特征。A 、四种 CMS 亚型结肠直肠癌病例的轨迹。B 、热图和 K 均值显示四种 CMS 亚型的动态波。C 、热图显示四种 CMS 亚型的临床特征、突变、臂/峰值事件和免疫浸润(Fisher 精确检验)。D 、 chr10q23.21 增益(左)和TP53突变(右)的轨迹。E 、四种 CMS 亚型在结肠直肠癌进展中的致癌途径简要总结。*,P < 0.05;**, P < 1.0E-2;***, P < 1.0E-3;****, P < 1.0E-4;ns,不显著,> 0.05。
为了分析 MSI 和 MSS 之间的差异(图 7),我们使用了 MSI 和 MSS 肿瘤之间的 DEP(Wilcoxon 秩和检验,FDR < 0.05,MSI 与 MSS 比率 ≥ 2 或 ≤ 0.5),然后应用 GSEA 进行通路富集。
图 7.
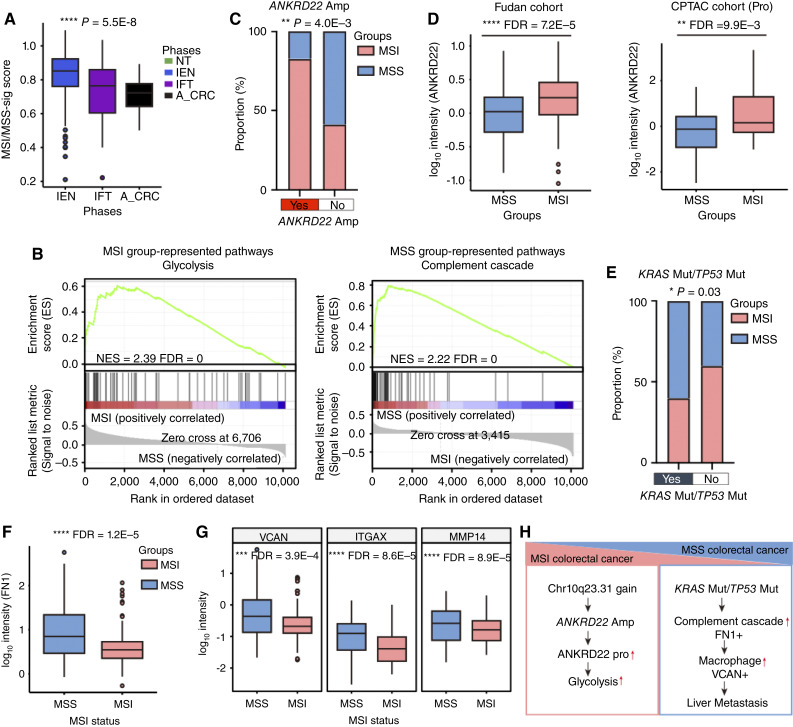
根据 MSI 状态对结直肠癌进展的分化。A ,箱线图显示结直肠癌进展中的 MSI/MSS-sig 评分(Kruskal-Wallis 检验)。B , MSI 组与 MSS 组相比,糖酵解(左)和补体级联途径(右)的 GSEA 图(KEGG 基因集)。C ,ANKRD22扩增组和 WT 组结直肠癌进展中的 MSI 比例(Fisher 精确检验)。D ,箱线图显示复旦队列和 CPTAC 队列中 MSI 组与 WT 组相比 ANKRD22 在蛋白质水平上的表达情况。(Wilcoxon 秩和检验)。E ,复旦队列和 MSKCC 队列中KRAS和TP53突变在结直肠癌进展中的比例(Fisher 精确检验)。F和G,箱线图显示MSI 组和 MSS 组中FN1( F )和 VCAN、ITGAX 和 MMP14的表达(Wilcoxon 秩和检验; G)。H ,基于 MSI 状态的结肠直肠癌特征简要总结。误差线,平均值 ± SEM。*,P < 0.05;**, P < 1.0E-2;***, P < 1.0E-3;****, P < 1.0E-4;ns,无显著性,> 0.05。
为了分析AOM/DSS导入结直肠癌小鼠模型三组(对照组、第I周期组、第II周期组)的分子特征(图8 ),采用各组高表达蛋白(Kruskal-Wallis检验,FDR < 0.05),然后使用ConsensusPathDB( http://cpdb.molgen.mpg.de/ )进行富集。
图 8.
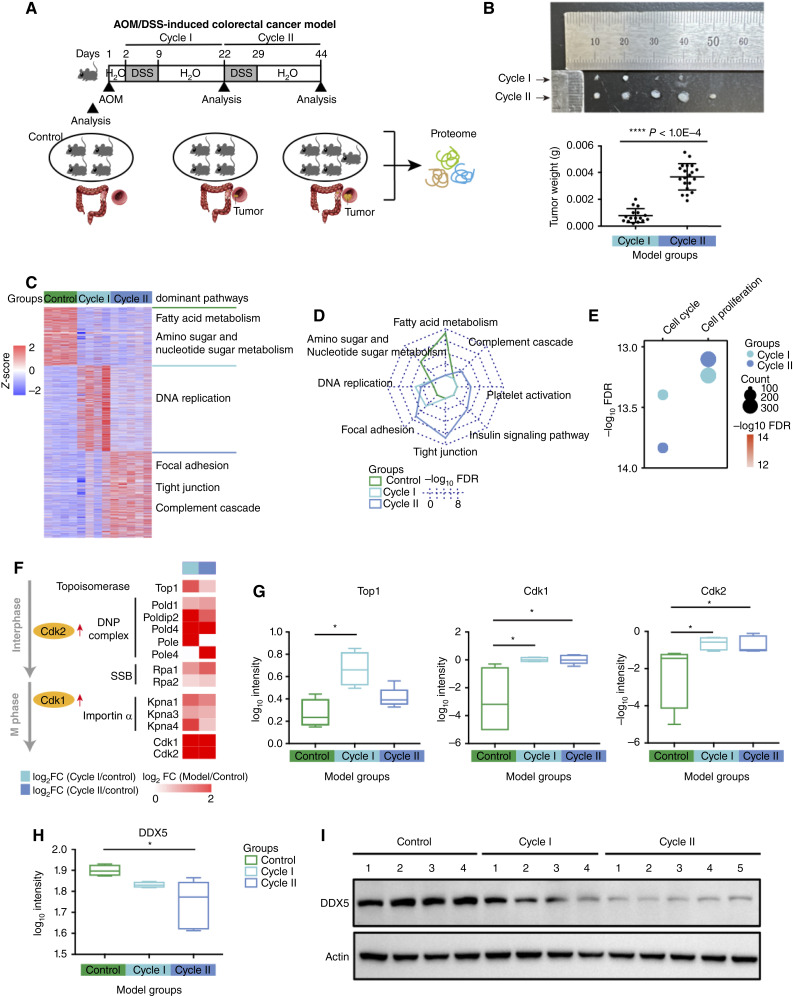
结肠直肠癌致癌小鼠模型的蛋白质组学特征。A , AOM/DSS 诱导的结肠直肠癌致癌小鼠模型示意图。B , AOM/DSS 诱导的结肠直肠癌小鼠模型的肿瘤重量(t检验)。C ,热图显示三组中的 DEP 和相关的主要生物学途径。D ,蜘蛛图显示三组中的代表途径。E ,周期 I 和 II 组中的主要途径。F ,周期 I 和 II 组中细胞周期和细胞增殖相关蛋白的表达。G ,箱线图显示三组中 Top1、Cdk1 和 Cdk2 的表达(Wilcoxon 秩和检验;Kruskal-Wallis 检验)。H ,三组中 DDX5 的蛋白质水平(Kruskal-Wallis 检验)。I ,蛋白质印迹显示 AOM/DSS 诱导的结肠直肠癌小鼠模型的肿瘤组织中 DDX5 的表达。误差线,平均值±SEM。 *,P < 0.05;**, P < 1.0E−2;***, P < 1.0E−3;****, P < 1.0E−4;ns,不显著,> 0.05。
构建和验证预测模型以区分KRAS Mut/ TP53 Mut 与其他、结直肠癌肝转移与 WT
使用 R 软件 v3.5.1,基于 20 种蛋白采用二项 Logistic 回归分析构建区分KRAS Mut/ TP53 Mut 与其他、结直肠癌肝转移与 WT 的预测模型。采用后向逐步法进行特征选择。将样本随机分为训练集和测试集。此外,使用 ROC 分析(pROC R 包版本 1.16.2 和 caret R 包版本 6.0-86)验证该模型的诊断价值。使用灵敏度、特异度、准确度和 AUC 确定预测值。在验证队列中对预测模型进行验证。
补体级联和细胞外基质信号传导评分
使用 R 包 GSVA,利用单样本 GSEA 根据蛋白质组学数据为每个样本获得分数。使用 Pearson 相关性确定基质分数与补体级联/细胞外基质 (ECM) 信号之间的相关性。使用 R 包 GSVA 中实现的单样本 GSEA 执行推断的补体级联和 ECM 信号分数。
激酶活性预测和磷酸肽分析
使用 MaxQuant(RRID:SCR_014485)在同一数据库中搜索 101 份结直肠癌样本的磷酸化蛋白质组数据。如我们之前的研究(16、17)所述,将S 或 T 或 Y 的磷酸化设置为可变修饰,其中允许 3 次错误切割,光谱匹配的最低 Andromeda 评分为 40。所有样品中已鉴定的磷酸化位点的比例用于通过 KSEA 算法估算激酶活性。激酶-底物关系的信息来自公开数据库,包括 PhosphoSite(RRID:SCR_001837)、Phospho.ELM(RRID:SCR_001109)和 PhosphoPOINT(RRID:SCR_002109)。底物基序的信息来自文献或使用 Motif(sP)分析 KSEA 数据集获得。激酶-底物-基序网络分析参考自 PhosphoSitePlus ( https://www.phosite.org/homeAction , RRID: SCR_001837) 和 NetworKIN 3.0 (RRID: SCR_007818)。使用 R (版本 3.5.1) 中的 Kruskal–Wallis 检验进行统计分析。
轨迹推理方法和进展路径分析
我们利用单片眼镜(版本 2.10.1)和轨迹推断方法追踪 148 名早期结直肠癌患者的谱系。如我们之前的研究(16、17)所述,突出显示并筛选了平均表达量超过 1.0E−1 的蛋白质。使用 R(版本3.5.1)中 Rtsne(版本 0.15)的 Barnes–Hut 实现对数据集进行聚类并通过 t 分布随机邻域嵌入进行预处理。每位早期结直肠癌患者的所有阶段都被视为伪时间,以构建基于 CMS 和 CRIS 的亚型分类中结直肠癌患者的轨迹。
单元格
HEK293T 细胞系 (Cat# CRL-11268, RRID: CVCL_QW54)、HCT116 细胞系 (Cat# CCL-247, RRID: CVCL_0291)、SW480 细胞系 (Cat# CCL-228, RRID: CVCL_0546) 和 SW620 细胞系 (Cat# CCL-227, RRID: CVCL_0547) 均购自 ATCC。HEK293T 细胞、HCT116 细胞、SW480 细胞和 SW620 细胞在 DMEM/高葡萄糖培养基 (HyClone) 中培养,培养基中添加 10% FBS (BI)、100 U/mL 青霉素和 100 μg/mL 链霉素 (Sangon Biotech)。所有细胞均在 37°C 的加湿培养箱中培养,培养液中含有 5% CO 2。通过短串联重复分析(Cell ID,Promega)确认细胞系的遗传身份,最后于 2023 年 12 月重复一次。使用Venor GeM Kit(Minerva Biolabs)定期检测细胞是否感染支原体,所有细胞系的支原体污染检测结果均为阴性。
IHC 分析
2008 年 6 月至 2018 年 12 月,上海交通大学医学院新华医院收集了用于组织阵列的人类结直肠癌样本。所有样本收集均获得了机构审查委员会的批准和知情同意(n = 244)。根据一般方案对组织阵列进行 IHC 染色,使用 DDX5 兔抗体(Abcam,目录号 ab126730,RRID:AB_11130291,稀释度 1:250)分析 DDX5 的表达。通过染色强度和染色百分比对 IHC 的半定量分析进行评分。
逆转录病毒的包装和感染
为了生成针对人类 DDX5 过度表达的逆转录病毒,将目标序列克隆到 pBABE-FLAG-puro 载体中。为了生成针对人类 DDX5 的逆转录病毒 shRNA 构建体,将目标序列克隆到 pMKO.1 puro 载体(Addgene,Cat# 8452,RRID:Addgene_8452)中。shRNA 序列如下:
shDDX5:5ʹ-AGGTGGAAACATACAGAAGAA-3ʹ
细胞增殖
将细胞接种于 96 孔板中,密度为每孔 4,000 个细胞(HCT116 细胞、SW480 细胞和 SW620 细胞)。根据制造商的方案,使用 Cell Counting Kit-8(Dojindo)测定细胞数。简而言之,将培养基替换为 100 μL 新鲜培养基,其中含有 10 μL CCK8。在 37°C 下孵育 3 小时后,将培养板摇动 5 分钟,并在 450 nm 处读取光密度值。
小鼠异种移植体外研究
裸鼠(4-6 周龄,雄性,n = 32)来自中国上海 SLAC 实验室动物有限责任公司。所有小鼠程序均经新华医院动物护理和使用委员会批准(XHEC-NSFC-2021-326)。将 pMKO-Control、pMKO-shDDX5、pBABE-Control 和 pBABE-oeDDX5 细胞(1 × 10 6)分别悬浮在 100 mL 1 × PBS 中,然后皮下注射到双侧腋窝脂肪垫中。21 天后,通过脱颈法处死小鼠。解剖肿瘤、成像并称重。收集肿瘤组织,在 10% 中性缓冲福尔马林中固定,以备后续分析。
AOM/DSS诱发的小鼠结直肠癌模型
小鼠称重后,第 1 天腹腔注射 AOM(6-8 周龄,10 mg kg −1体重,溶于 PBS),然后从第 2 天开始,以 1.25%–1.5%–1.75% 的浓度逐步增加的方式,进行 3 次 DSS 溶解在饮用水中。在每个 DSS 周期中,小鼠饮用 DSS 水 7 天,然后在下一个周期前停止 14 天。在本研究中,小鼠在第 21 天(周期 I,n = 4)和第 44 天(周期 II,n = 5)处死。纵向解剖结肠后计算并拍照。具体而言,周期 I 和周期 II 中的肿瘤代表早期和晚期的样本。所有小鼠(n = 13)均饲养在无病原体动物设施(新华医院动物护理和使用委员会,XHEC-NSFC-2021-326)的通风笼中,可自由饮水和食用标准啮齿动物饮食。小鼠在受控温度(22°±2°)和 12 小时明暗循环下饲养。
免疫沉淀
对于免疫沉淀,用含有 50 mmol/L Tris-HCl(pH 7.5)、150 mmol/L NaCl 和多种蛋白酶抑制剂(苯甲基磺酰氟 1 mmol/L、抑肽酶 1 mg/mL、亮抑酶肽 1 mg/mL、胃酶抑素 1 mg/mL、Na 3 VO 4 1 mmol/L 和 NaF 1 mmol/L(用于乙酰化实验;另外还有 TSA 2.5 mmol/L 和 NAM 25 mmol/L,最终浓度为)的 1% Nonidet P40 缓冲液裂解细胞。将细胞裂解物与抗 FLAG M2 磁珠(Sigma)在 4°C 下孵育。用 NP40 缓冲液洗涤免疫复合物三次。然后使用指示的一抗检查裂解物和免疫沉淀物。
使用的网站和下载的蛋白质
在本研究中,为了下载巨噬细胞标志物(图 4),我们进入了 CellMarker 网站(http://xteam.xbio.top/CellMarker/download.jsp ),并通过筛选出关键词“巨噬细胞”下载了巨噬细胞相关标志物数据库(n = 285)。为了获得锚定蛋白,我们进入了人类蛋白质图谱(HPA; n = 49;https://www.proteinatlas.org ),并通过筛选出关键词“锚定蛋白”下载了锚定蛋白数据库(n = 49)。
量化和统计分析
所有数据均使用 R 和 GraphPad Prism 9 软件进行分析和绘图,并使用 R(版本 3.5.1)进行 Fisher 精确检验、Bartlett 检验、Kruskal-Wallis 检验、Wilcoxon 符号秩检验和 Spearman 相关性检验。条形图中的数据以平均值±SEM 表示。所有统计检验均为双尾,当(调整后的)P值 < 0.05 时认为具有统计学意义,使用 Benjamini-Hochberg 程序进行调整。Kaplan-Meier 图(对数秩检验)用于描述总体生存率。随机选择结直肠癌患者的组织样本。为了验证本研究中的结果,每个实验至少独立重复三次。所有实验均可靠地重现并在图例、方法和材料以及结果中指明。箱线图中的数据表示为中位数(中心线)、上四分位数和下四分位数(箱界)和 1.5× IQR(须)。对于样品处理、PCA 和共识聚类分析,所有研究人员都对结果不知情。
(责任编辑:佳学基因)