【佳学基因检测】唇腭裂会不会遗传基因检测
唇腭裂会不会遗传基因检测
基因检测可以帮助分析唇腭裂的遗传因素。唇腭裂是一种出生缺陷,其发生涉及多种遗传因素的复杂相互作用。基因检测可以帮助确定个体是否携带与唇腭裂相关的遗传变异或突变,从而评估其遗传风险和患病可能性。
具体来说,基因检测在以下几个方面对唇腭裂的研究和临床实践有重要意义:
遗传风险评估:通过分析相关基因的变异或突变,可以评估个体患唇腭裂的遗传风险。某些基因变异如IRF6、PAX9等已被证实与唇腭裂的发生有关。
家族遗传:基因检测有助于识别家族中存在的遗传性唇腭裂的风险,帮助家庭进行遗传咨询和筛查。
研究和进展:基因检测为科学研究提供了重要数据支持,促进了对唇腭裂遗传机制的深入理解和新治疗方法的发展。
综上所述,基因检测可以通过分析相关的遗传变异和突变,帮助理解唇腭裂的遗传基础,并为个体化的遗传风险评估提供支持。
唇腭裂致病基因鉴定过程
唇腭裂的致病基因鉴定基因解码通过GWAS 已发现 43 个与 非综合征性颌面裂 (NSOFC) 相关的基因/位点,但大多数先前研究使用的是 CL/P 亚型的混合样本,而不是单独的 腭裂(CPO)或 唇裂(CLO)。唇腭裂致病基因鉴定基因解码的研究结果使用基于 腭裂(CPO)和 唇裂(CLO) GWAS 数据的遗传因子及其功能本体来定义 腭裂(CPO)和 唇裂(CLO) 亚型。在本研究中,唇腭裂致病基因鉴定基因解码确定了 18 个有助于 CPO、唇裂(CLO) 或 CLP 的基因/位点。其中 14 个是新发现的,12 个含有发育转录因子 (TF),这表明 TF 是 非综合征性颌面裂 (NSOFC) 亚型发病的关键因素。唇腭裂致病基因鉴定基因解码在与 腭裂(CPO)和 唇裂(CLO) 最相关的位点IRF6中观察到了相反的作用;以前的 CL/P GWAS 忽略了此信息。此外,唇腭裂致病基因鉴定基因解码发现IRF6的基因表达剂量在驱动 腭裂(CPO)或 唇裂(CLO) 方面起着重要作用。此外,唇腭裂致病基因鉴定基因解码发现PAX9是 腭裂(CPO)的一个强遗传因子。这些结果表明转录因子是 非综合征性颌面裂 (NSOFC) 亚型发病的关键遗传原因。
典型的 GWAS 和重复研究确定了 9 个导致 非综合征性颌面裂 (NSOFC) 的新位点
为了鉴定 腭裂(CPO)或 唇裂(CLO) 特有的 非综合征性颌面裂 (NSOFC) 易感基因/位点,唇腭裂致病基因鉴定基因解码使用 Illumina HumanOmniZhongHua-8 BeadChip对 935 名无关 腭裂(CPO)患者、948 名无关 唇裂(CLO) 患者和 5,050 名无关南方汉族对照个体进行了基因分型,该芯片在发现阶段有 900,015 个单核苷酸多态性 (SNP)。队列样本收集如图所示表1。数据集的总基因分型率为 0.9949。
表1:各研究组的样本数
| 群组 | CPL | CLO | CLP | 对照组 | 基因型方法 |
|---|---|---|---|---|---|
| 发现(南方汉族,第一群体) | 930 | 945 | 5068 | HumanOmniZhongHua-8 微珠芯片 (Illumina) | |
| 复制(南方汉族,第二组) | 724 | 781 | 3265 | Sequenom Mass ARRAY 系统 | |
| 复制(北方汉族,第 3 组) | 417 | 492 | 1832 | Sequenom Mass ARRAY 系统 | |
| 复制(南方汉族,第 4 组) | 2270 | 3265 | Sequenom Mass ARRAY 系统 | ||
| 复制(北方汉族,第五组) | 427 | 1832 | Sequenom Mass ARRAY 系统 | ||
| 总数 | 2071 | 2218 | 2697 | 10165 |
对参与者和 SNP 进行标准质量对照过滤,并通过群体分层分析排除质量差和遗传异质性的样本后,唇腭裂致病基因鉴定基因解码获得了 930 名单独的 腭裂(CPO)患者、945 名单独的 唇裂(CLO) 患者和 5,048 名对照个体的基因型数据(表 2,发现队列)。主成分分析(PCA)结果表明,其余病例和对照在遗传上匹配良好,没有明显的人口分层迹象。发现队列中的基因组膨胀因子(λ GC)为 腭裂(CPO)1.016 和 唇裂(CLO) 1.031,这表明关联检验统计数据并未受到人口子结构的显著影响。唇腭裂致病基因鉴定基因解码使用 PLINK 1.9 版软件,采用逻辑斯蒂检验对性别和 PC(C1、C2、C3 和 C4)进行了 GWAS 分析。为了进一步增加基因组覆盖率,唇腭裂致病基因鉴定基因解码进行了归因分析以推断其他常见 SNP 的基因型(见方法)。P值的曼哈顿图(使用 R 包“qqman”)显示在图1腭裂(CPO)在发现阶段没有表现出强的关联信号。如果唇腭裂致病基因鉴定基因解码使用正常的 GWAS 显著性截断值,比如P < 5 × 10 −8,唇腭裂致病基因鉴定基因解码可能会在第一次重复实验中遗漏一些真正的关联基因。腭裂(CPO)的关联会很弱,因为以前基于三重奏的 腭裂(CPO)GWAS 分析没有发现很多 腭裂(CPO)主效应基因/基因座。之前的研究表明 腭裂(CPO)的遗传模型应该由许多次要效应基因/基因座组成。因此,唇腭裂致病基因鉴定基因解码在发现阶段使用了P < 9 × 10 −7截断值进行第一轮重复的 SNP 选择。共有 22 个基因座在 GWAS 发现阶段显示出与 腭裂(CPO)或 唇裂(CLO) 显著关联的证据(即超过P < 9 × 10 −7)。
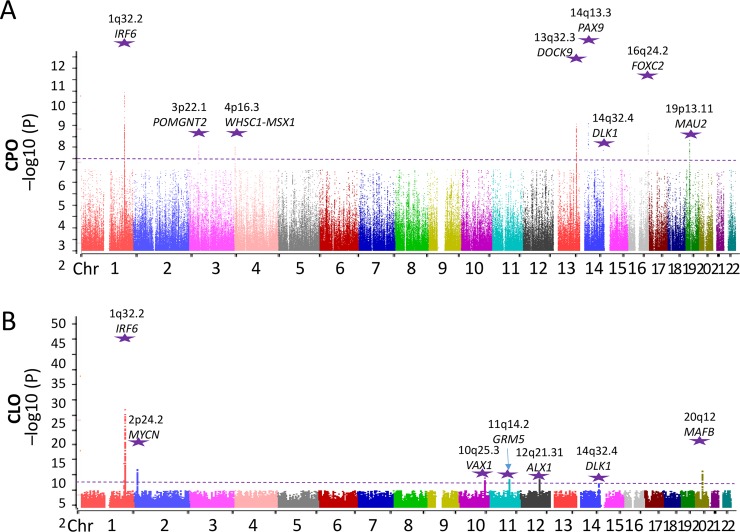
图1:典型 GWAS 识别的基因/基因座的曼哈顿图
曼哈顿图显示了通过 Hardy–Weinberg 检验的 SNP 关联分析的−log 10 P值, P值 > 1.0 × 10 −6。该图包括发现队列的 GWAS 数据,包括 930 名 腭裂(CPO)患者(A)、945 名 唇裂(CLO) 患者(B)和 5048 名中国南方汉族健康对照个体,根据常染色体上 SNP 的相应位置绘制。紫色虚线显示本研究的外显子组显着性阈值(P = 9 × 10 −7)。紫色星号表示最终荟萃分析的结果,该分析结合了 GWAS 发现队列和两个重复队列,总共包括 2071 例 腭裂(CPO)病例、2218 例 唇裂(CLO) 病例和 10165 名对照个体。
表 2:通过典型的 GWAS 识别的 非综合征性颌面裂 (NSOFC) 基因/基因座。
通过对发现和复制结果的荟萃分析,对 腭裂(CPO)或 唇裂(CLO) 中在多重检验校正水平(P < 5 × 10 −8 )上显著的 SNP 进行典型的 GWAS 结果。
| 单核苷酸多态性 | 基因座 | 受影响的基因(a) | 等位基因 | 群组 | 发现(c)N = 6943 | 复制 CHS (d) N = 7040 | 复制 CHB (e) N = 3163 | 合计(f)N = 17151 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| MAF (b) | P | OR | MAF (b) | OR | 或者 | MAF (b) | P | OR | P | OR | I(g) | ||||||||
| 新风险基因位点标记的关联结果 (n = 9) | |||||||||||||||||||
| rs6791526 | 3p22.1 |
|
T/C | CPO | 0.107/0.073 | 6.23 × 10 −7 | 1.53 | 0.093/0.068 | 2.13 × 10 −3 | 1.40 | 0.086/0.063 | 1.43 × 10 −2 | 1.4 | 1.62 × 10 −10 | 1.46 | 0 | |||
| CLO | 0.083/0.073 | 0.129 | 1.15 | 0.081/0.068 | 7.19 × 10 −2 | 1.21 | 0.085/0.063 | 1.37 × 10 −2 | 1.39 | 1.39 × 10 −4 | 1.22 | 0 | |||||||
| CLP | 0.078/0.068 | 3.41 × 10 −2 | 1.16 | 0.060/0.063 | 0.796 | 1.047 | 5.8 × 10 −2 | 1.14 | 0 | ||||||||||
| rs3468 | 4p16.3 | WHSC1 | G/A | CPO | 0.522/0.455 | 8.28 × 10 −7 | 1.33 | 0.499/0.454 | 1.24 × 10 −3 | 1.20 | 0.496/0.455 | 8.73 × 10 −2 | 1.18 | 5.4 × 10 −11 | 1.256 | 20.56 | |||
| CLO | 0.451/0.455 | 0.7388 | 0.99 | 0.471/0.454 | 0.252 | 1.07 | 0.455/0.455 | 0.714 | 1.00 | 0.57 | 1.02 | 0 | |||||||
| CLP | 0.474/0.454 | 5.61 × 10 −2 | 1.083 | 0.460/0.455 | 0.803 | 1.02 | 5.52 × 10 −2 | 1.070 | 0 | ||||||||||
| rs57700751 | 11q14.2 | GRM5 | C/T | CPO | 0.069/0.068 | 0.87 | 1.02 | 0.061/0.055 | 0.43 | 1.12 | 0.062/0.071 | 0.403 | 0.868 | 0.79 | 1.01 | 0 | |||
| CLO | 0.101/0.068 | 4.75 × 10 −7 | 1.547 | 0.069/0.055 | 3.92 × 10 −2 | 1.287 | 0.089/0.071 | 9.2 × 10 −2 | 1.27 | 3.0 × 10 −8 | 1.40 | 16.4 | |||||||
| CLP | 0.072/0.055 | 5.08 × 10 −4 | 1.335 | 0.073/0.071 | 0.347 | 1.024 | 1.2 × 10 −3 | 1.25 | 60.23 | ||||||||||
| rs765366 | 12q21.31 | ALX1 | G/A | CPO | 0.185/0.168 | 7.1 × 10 −2 | 1.125 | 0.144/0.136 | 0.786 | 1.073 | 0.169/0.163 | 0.786 | 1.040 | 5.8 × 10 −2 | 1.09 | 0 | |||
| CLO | 0.216/0.168 | 5.26 × 10 −7 | 1.37 | 0.156/0.136 | 4.84 × 10 −2 | 1.174 | 0.189/0.163 | 6× 10−2 | 1.19 | 4.30 × 10 −8 | 1.27 | 30.0 | |||||||
| CLP | 0.145/0.136 | 0.181 | 1.08 | 0.143/0.163 | 0.831 | 0.86 | 0.58 | 1.027 | 77.66 | ||||||||||
| rs4646211 | 13q32.3 | DOCK9 | C/T | CPO | 0.423/0.346 | 2.82 × 10 −10 | 1.38 | 0.333/0.296 | 8.71 × 10 −3 | 1.184 | 0.314/0.273 | 2.84 × 10 −2 | 1.22 | 4.78 × 10 −12 | 1.28 | 50 | |||
| CLO | 0.348/0.346 | 0.495 | 1.00 | 0.315/0.296 | 0.181 | 1.091 | 0.273/0.273 | 0.11 | 1.00 | 0.42 | 1.03 | 0 | |||||||
| CLP | 0.311/0.296 | 0.11 | 1.07 | 0.259/0.273 | 1.11 | 0.928 | 0.28 | 1.04 | 54.32 | ||||||||||
| rs730643 | 14q13.3 | PAX9 | G/A | CPO | 0.263/0.338 | 2.84 × 10 −8 | 0.70 | 0.326/0.388 | 1.26 × 10 −5 | 0.76 | 0.314/0.363 | 8.90 × 10 −3 | 0.804 | 2.92 × 10 −16 | 0.74 | 7.63 | |||
| CLO | 0.334/0.338 | 0.863 | 0.98 | 0.372/0.388 | 0.226 | 0.93 | 0.363/0.363 | 0.792 | 1.00 | 0.32 | 0.97 | 0 | |||||||
| CLP | 0.377/0.388 | 0.287 | 0.96 | 0.382/0.363 | 0.3 | 1.083 | 0.68 | 0.983 | 41.14 | ||||||||||
| rs2415363 | 14q13.3 | PAX9 | A/C | CPO | 0.263/0.338 | 2.84 × 10 −8 | 0.70 | 0.326/0.385 | 2.17 × 10 −5 | 0.77 | 0.324/0.363 | 7.03 × 10 −2 | 0.841 | 2.76 × 10 −14 | 0.75 | 44.52 | |||
| CLO | 0.334/0.338 | 0.863 | 0.98 | 0.356/0.385 | 4.56 × 10 −2 | 0.88 | 0.36/0.363 | 0.896 | 0.987 | 0.11 | 0.75 | 10.89 | |||||||
| CLP | 0.380/0.385 | 0.652 | 0.97 | 0.385/0.363 | 0.241 | 1.101 | 0.91 | 1.00 | 51.74 | ||||||||||
| rs60708031 | 14q13.3 | PAX9 | T/G | CPO | 0.263/0.338 | 9.29 × 10 −8 | 0.70 | 0.323/0.384 | 1.70 × 10 −5 | 0.76 | 0.317/0.360 | 2.03 × 10 −2 | 0.825 | 4.39 × 10 −15 | 0.75 | 29.66 | |||
| CLO | 0.334/0.338 | 0.932 | 0.983 | 0.363/0.384 | 0.136 | 0.915 | 0.360/0.360 | 0.764 | 1.00 | 0.28 | 0.96 | 0 | |||||||
| CLP | 0.392/0.384 | 0.387 | 1.035 | 0.388/0.360 | 0.173 | 1.125 | 0.28 | 0.996 | 0 | ||||||||||
| rs730570 | 14q32.2 | DLK1 | A/G | CPO | 0.243/0.197 | 1.71 × 10 −5 | 1.309 | 0.240/0.204 | 3.32 × 10 −3 | 1.228 | 0.225/0.181 | 3.32 × 10 −3 | 1.34 | 6.59 × 10 −10 | 1.28 | 0 | |||
| CLO | 0.248/0.197 | 6.14 × 10 −7 | 1.344 | 0.233/0.204 | 1.50 × 10 −2 | 1.18 | 0.206/0.181 | 8.70 × 10 −2 | 1.18 | 2.53 × 10 −8 | 1.253 | 22.1 | |||||||
| CLP | 0.219/0.204 | 7.84 × 10 −2 | 1.09 | 0.159/0.181 | 0.184 | 0.856 | 0.31 | 1.045 | 77.77 | ||||||||||
| rs72812203 | 16q24.2 | FOXC2-FOXL1 | G/T | CPO | 0.276/0.213 | 8.90 × 10 −8 | 1.40 | 0.239/0.213 | 2.83 × 10 −2 | 1.16 | 0.237/0.201 | 2.83 × 10 −2 | 1.23 | 4.2× 10 −10 | 1.28 | 56.83 | |||
| CLO | 0.253/0.213 | 1.13 × 10−3 | 1.174 | 0.237/0.213 | 3.88 × 10−2 | 1.15 | 0.189/0.201 | 0.646 | 1.00 | 2.4× 10−3 | 1.129 | 14.61 | |||||||
| CLP | 0.221/0.213 | 0.321 | 1.046 | 0.189/0.201 | 0.646 | 0.924 | 0.63 | 1.13 | 24.41 | ||||||||||
| rs8061677 | 16q24.2 | FOXC2-FOXL1 | C/T | CPO | 0.242/0.205 | 5.79 × 10−8 | 1.40 | 0.234/0.207 | 2.10 × 10−2 | 1.17 | 0.231/0.196 | 2.10 × 10−2 | 1.29 | 9.11 × 10−11 | 1.29 | 48.84 | |||
| 唇裂(CLO) | 0.241/0.205 | 1.18 × 10−4 | 1.23 | 0.235/0.207 | 1.52 × 10−2 | 1.180 | 0.196/0.196 | 0.951 | 0.991 | 2.3 × 10−4 | 1.16 | 50.49 | |||||||
| CLP | 0.226/0.207 | 1.52 × 10−2 | 1.12 | 0.194/0.196 | 0.951 | 0.991 | 3.7 × 10−2 | 1.09 | 23.25 | ||||||||||
| rs1009136 | 19p13.11 | MAU2 | A/G | CPO | 0.359/0.295 | 8.84 × 10−7 | 1.34 | 0.341/0.311 | 2.90 × 10−2 | 1.14 | 0.330/0.288 | 1.52 × 10−2 | 1.21 | 2.66 × 10−9 | 1.25 | 46.17 | |||
| 唇裂(CLO) | 0.309/0.295 | 2.2 × 10−2 | 1.069 | 0.307/0.311 | 0.721 | 0.98 | 0.289/0.288 | 0.84 | 1.00 | 0.51 | 1.02 | 0 | |||||||
| CLP | 0.295/0.311 | 7.03 × 10−2 | 0.927 | 0.295/0.288 | 0.689 | 1.035 | 0.27 | 0.95 | 22.4 | ||||||||||
| Association results for markers from the replicated risk loci (n = 4) | |||||||||||||||||||
| rs2235371 | 1q32.2 | IRF6 | T/C | CPO | 0.472/0.405 | 3.49 × 10−7 | 1.314 | 0.471/0.390 | 2.07 × 10−6 | 1.394 | 0.373/0.378 | 0.819 | 0.9817 | 1.90 × 10−9 | 1.254 | 33.72 | |||
| 唇裂(CLO) | 0.268/0.405 | 1.44 × 10−26 | 0.5387 | 0.273/0.390 | 1.78 × 10−13 | 0.588 | 0.242/0.378 | 6.25 × 10−13 | 0.528 | 1.25 × 10−48 | 0.551 | 0 | |||||||
| CLP | 0.319/0.390 | 2.49 × 10−12 | 0.7335 | 0.263/0.378 | 1.79 × 10−9 | 0.5886 | 4.50 × 10−19 | 0.702 | 39.68 | ||||||||||
| rs2235377 | 1q32.2 | IRF6 | C/T | CPO | 0.483/0.412 | 7.98 × 10−8 | 1.333 | 0.479/0.392 | 2.16 × 10−4 | 1.426 | 0.479/0.384 | 2.16 × 10−4 | 1.43 | 1.02 × 10−13 | 1.368 | 0 | |||
| 唇裂(CLO) | 0.273/0.412 | 2.89 × 10−27 | 0.5356 | 0.275/0.392 | 8.91 × 10−7 | 0.589 | 0.250/0.384 | 7.55 × 10−6 | 0.535 | 3.70 × 10−36 | 0.546 | 0 | |||||||
| CLP | 0.225/0.392 | 2.89 × 10−27 | 0.5356 | 0.251/0.384 | 7.55 × 10−6 | 0.535 | 4.23 × 10−31 | 0.536 | 0 | ||||||||||
| rs12405750 | 1q32.2 | IRF6 | T/C | CPO | 0.483/0.412 | 7.98 × 10−8 | 1.333 | 0.480/0.393 | 3.47 × 10−7 | 1.427 | 0.389/0.387 | 0.985 | 1.001 | 8.80 × 10−11 | 1.276 | 33.91 | |||
| 唇裂(CLO) | 0.272/0.412 | 1.82 × 10−27 | 0.5341 | 0.278/0.393 | 1.29 × 10−12 | 0.6014 | 0.249/0.387 | 2.92 × 10−13 | 0.526 | 8.13 × 10−49 | 0.553 | 1.66 | |||||||
| CLP | 0.321/0.393 | 1.21 × 10−12 | 0.7304 | 0.268/0.387 | 6.22 × 10−10 | 0.5819 | 9.56 × 10−20 | 0.698 | 31.14 | ||||||||||
| rs10863790 | 1q32.2 | IRF6 | C/A | CPO | 0.481/0.411 | 1.47 × 10−7 | 1.325 | 0.483/0.387 | 5.04 × 10−5 | 1.481 | 0.379/0.387 | 0.762 | 0.9723 | 1.20 × 10−8 | 1.27 | 32.47 | |||
| 唇裂(CLO) | 0.272/0.411 | 1.30 × 10−27 | 0.5331 | 0.279/0.387 | 1.13 × 10−7 | 0.611 | 0.246/0.387 | 1.30 × 10−9 | 0.52 | 1.16 × 10−40 | 0.548 | 0 | |||||||
| CLP | 0.321/0.387 | 5.51 × 10−10 | 0.749 | 0.2676/0.387 | 7.00 × 10−10 | 0.5823 | 7.64 × 10−17 | 0.709 | 34.24 | ||||||||||
| rs72741048 | 1q32.2 | IRF6 | T/A | CPO | 0.588/0.506 | 8.64 × 10−10 | 1.392 | 0.588/0.510 | 8.18 × 10−7 | 1.36 | 0.5158/0.493 | 0.321 | 1.094 | 3.07 × 10−15 | 1.314 | 48.33 | |||
| 唇裂(CLO) | 0.368/0.506 | 9.56 × 10−26 | 0.5665 | 0.37/0.510 | 1.39 × 10−8 | 0.612 | 0.352/0.493 | 2.85 × 10−9 | 0.558 | 8.22 × 10−40 | 0.575 | 0 | |||||||
| CLP | 0.418/0.510 | 9.43 × 10−11 | 0.749 | 0.3937/0.493 | 3.91 × 10−7 | 0.664 | 5.20 × 10−16 | 0.728 | 10.62 | ||||||||||
| rs189000 | 1q32.2 | IRF6 | A/G | CPO | 0.349/0.419 | 9.55 × 10−8 | 0.7438 | 0.341/0.408 | 9.62 × 10−5 | 0.752 | 0.388/0.410 | 0.237 | 0.9099 | 2.28 × 10−10 | 0.782 | 17.32 | |||
| 唇裂(CLO) | 0.499/0.419 | 1.14 × 10−9 | 1.381 | 0.505/0.408 | 1.91 × 10−9 | 1.484 | 0.476/0.410 | 7.56 × 10−4 | 1.3 | 1.17 × 10−19 | 1.395 | 0 | |||||||
| CLP | 0.468/0.408 | 6.81 × 10−9 | 1.279 | 0.476/0.410 | 7.97 × 10−4 | 1.31 | 2.39 × 10−11 | 1.285 | 0 | ||||||||||
| rs1387934 | 1q32.2 | IRF6 | T/C | CPO | 0.349/0.418 | 1.17 × 10−7 | 0.7453 | 0.342/0.409 | 9.25 × 10−5 | 0.752 | 0.391/0.411 | 0.298 | 0.9203 | 4.06 × 10−10 | 0.785 | 21.52 | |||
| 唇裂(CLO) | 0.500/0.418 | 6.95 × 10−10 | 1.387 | 0.506/0.409 | 2.12 × 10−9 | 1.482 | 0.476/0.411 | 7.45 × 10−4 | 1.3 | 7.47 × 10−20 | 1.397 | 0 | |||||||
| CLP | 0.468/0.409 | 1.16 × 10−8 | 1.274 | 0.479/0.411 | 4.58 × 10−4 | 1.32 | 2.54 × 10−11 | 1.284 | 0 | ||||||||||
| rs4832468 | 2p24.2 | MYCN | C/T | CPO | 0.2516/0.2913 | 9.63 × 10−4 | 0.818 | 0.233/0.281 | 2.46 × 10−2 | 0.777 | 0.233/0.288 | 2.46 × 10−2 | 0.777 | 5.38 × 10−6 | 0.803 | 0 | |||
| 唇裂(CLO) | 0.213/0.2913 | 3.24 × 10−11 | 0.6584 | 0.204/0.281 | 3.41 × 10−4 | 0.6525 | 0.201/0.288 | 1.88 × 10−3 | 0.624 | 5.21 × 10−16 | 0.653 | 0 | |||||||
| CLP | 0.178/0.282 | 3.24 × 10−12 | 0.6584 | 0.201/0.288 | 1.88 × 10−3 | 0.624 | 9.37 × 10−11 | 0.652 | 0 | ||||||||||
| rs7552 | 2p24.2 | MYCN | A/G | CPO | 0.254/0.292 | 1.39 × 10−3 | 0.8236 | 0.229/0.284 | 1.11 × 10−2 | 0.7488 | 0.229/0.290 | 1.11 × 10−2 | 0.749 | 2.52 × 10−6 | 0.796 | 0 | |||
| 唇裂(CLO) | 0.211/0.292 | 7.23 × 10−12 | 0.6489 | 0.199/0.284 | 1.01 × 10−4 | 0.625 | 0.204/0.290 | 3.32 × 10−3 | 0.628 | 6.44 × 10−17 | 0.642 | 0 | |||||||
| CLP | 0.178/0.284 | 3.23 × 10−12 | 0.648 | 0.204/0.290 | 3.32 × 10−3 | 0.628 | 8.25 × 10−14 | 0.645 | 0 | ||||||||||
| rs7902527 | 10q25.3 | VAX1 | A/G | CPO | 0.297/0.279 | 0.15 | 1.088 | 0.275/0.303 | 0.202 | 0.8724 | 0.275/0.287 | 0.202 | 0.8724 | 0.977 | 1.001 | 22.39 | |||
| 唇裂(CLO) | 0.346/0.279 | 3.21 × 10−8 | 1.366 | 0.335/0.303 | 4.0 × 10−2 | 1.23 | 0.301/0.287 | 0.619 | 1.07 | 3.15 × 10−8 | 1.273 | 10.25 | |||||||
| CLP | 0.194/0.303 | 3.21 × 10−8 | 1.366 | 0.301/0.287 | 0.619 | 1.07 | 1.25 × 10−7 | 1.318 | 23.82 | ||||||||||
| rs4752028 | 10q25.3 | VAX1 | C/T | CPO | 0.343/0.334 | 0.483 | 1.04 | 0.348/0.347 | 0.959 | 1.005 | 0.348/0.321 | 0.959 | 1.005 | 0.556 | 1.026 | 0 | |||
| 唇裂(CLO) | 0.408/0.334 | 3.62 × 10−9 | 1.378 | 0.395/0.347 | 3.90 × 10−2 | 1.23 | 0.367/0.32 | 0.113 | 1.23 | 2.58 × 10−10 | 1.328 | 0 | |||||||
| CLP | 0.450/0.347 | 3.62 × 10−9 | 1.378 | 0.367/0.32 | 0.113 | 1.23 | 1.55 × 10−9 | 1.354 | 0 | ||||||||||
| rs17820943 | 20q12 | MAFB | T/C | CPO | 0.434/0.456 | 0.105 | 0.9166 | 0.435/0.464 | 0.224 | 0.8878 | 0.476/0.482 | 0.776 | 0.9748 | 5.65 × 10−2 | 0.924 | 0 | |||
| 唇裂(CLO) | 0.370/0.456 | 6.95 × 10 −11 | 0.7015 | 0.372/0.464 | 0.40 × 10 −4 | 0.482 | 0.404/0.482 | 1.05 × 10 −3 | 0.729 | 2.08 × 10 −17 | 0.702 | 0 | |||||||
| CLP | 0.378/0.464 | 0.38 × 10 −14 | 0.482 | 0.385/0.482 | 1.02 × 10 −6 | 0.673 | 3.77 × 10 −20 | 0.695 | 0 | ||||||||||
(a)该区域最可能的易感基因
(b)MAF:受影响/不受影响
(c)930 CPO、945 唇裂(CLO) 和 5048 对照
(d)724 台 CPO、781 台 唇裂(CLO)、2,270 台 CLP 和 3,265 对照
(e)417 家 CPO、492 家 唇裂(CLO)、427 家 CLP 和 1,832 家对照
(f)共计 17,151 个样本:2,071 个 CPO、2,218 个 唇裂(CLO)、2,697 个 CLP 和 10,165 个对照
(g)I2异质性。CHS:中国南方人;CHN:中国北方人。
此外,之前报道的 22 个基因座(包括 32 个负责 CL/P、腭裂(CPO)或 CLP 的 SNP)也显示出与 腭裂(CPO)或 唇裂(CLO) 的边际关联( P < 0.05)。在这些基因座中,本研究中报道的两个 腭裂(CPO)SNPs rs61776460(1p36.11(GRHL3,P = 9.31× 10 −3))和 rs604328(5p13.2(UGT3A2,P = 3.15× 10 −3))与 腭裂(CPO)弱关联。基因解码报道的四个 腭裂(CPO)SNPs 。 ( CTNNA2中的 rs80004662 和 rs113691307 、 SULT2A1中的 rs62529857以及DACH1中的 rs2325377 ,唇腭裂致病基因鉴定基因解码无法在唇腭裂致病基因鉴定基因解码的芯片中找到这些 SNP。在本研究中,两个已报道的 CLP SNP 4q28.1 中的 rs908822(LOC285419,P = 2.34 × 10 −3 )和 8p11.23 中的 rs13317(BAG4/FGFR1,P = 4.21 × 10 −3)与 腭裂(CPO)弱关联。两个已报道的 CL/P SNP 8q24.21 中的 rs987525(AC068570 . 1,P = 5.53 × 10 −3)和 13q31.1 中的 rs60417080(RP11-501G7 . 1,P = 8.83 × 10 −3)在本研究中也与 腭裂(CPO)呈弱关联。已报道的 1p22.1–21.3 中的 CLP SNPs rs560426 和 rs66515264(ARHGAP29,分别为P = 1.79 × 10 −3和P = 4.83 × 10 −3),以及 8q21.3 中的 rs1034832(DCAF4L2/CTB-118P15 . 2,P = 2.60 × 10 −5)与 唇裂(CLO) 弱关联。在同一基因座中,已报道与 CL/P 相关的 rs12543318 也与 唇裂(CLO) 弱关联(P = 3.79 × 10 −5 )。另外已报道的 3 个 CL/P SNPs 为 16p13.3 中的 rs8049367(RP11-462G12 . 2 , P = 2.95 × 10 −4 )、17p13.1 中的 rs4791774(NTN1 , P = 5.87 × 10 −5 ) 和 17q22 中的 rs227731(NOG, P = 4.35 × 10 −4)在本研究中显示出与 唇裂(CLO) 的弱关联。
为了复制唇腭裂致病基因鉴定基因解码发现的队列中发现的关联,唇腭裂致病基因鉴定基因解码首先在 724 名 腭裂(CPO)患者、781 名 唇裂(CLO) 患者和 3,265 名对照个体(中国南方复制队列)中选择了上述 22 个发现基因座中的 48 个 SNP 用于复制分析。15 个基因座中的 32 个 SNP 与 腭裂(CPO)或 唇裂(CLO) 保持统计学上显着的关联(P < 0.05)。然后,唇腭裂致病基因鉴定基因解码在北方民族的二级复制队列中对这 32 个 SNP 进行了基因分型,其中包括 417 名无关的 腭裂(CPO)患者、492 名无关的 唇裂(CLO) 患者和 1,832 名无关的正常对照个体。
唇腭裂致病基因鉴定基因解码对这三个队列进行了荟萃分析,得出了与 腭裂(CPO)相关的八个基因/位点,其显著性超过了全基因组的统计学意义 ( P < 5.0 × 10 −8 ) (图 1A,图 2和表 2)八个基因/位点中的一个,IRF6,先前报道与CPO相关:IRF6(rs12405750,P = 8.8×10−11 )。同时,有 7 个基因/位点是新发现的:POMGNT2(rs6791526,P = 1.62 × 10 −10)、WHSC1(rs3468,P = 5.4 × 10 −11)、DOCK9(rs4646211,P = 4.78 × 10 −12)、PAX9(rs730643,P = 2.92 × 10 −16)、DLK1(rs730570,P = 6.59 × 10 −10)、FOXC2-FOXL1(rs72812203,P = 4.2 × 10 −10)和MAU2(rs1009136,P = 2.66 × 10 −9)。有 7 个基因/位点与 唇裂(CLO) 的关联超过全基因组统计学意义(图 1B,图 2和表 2);其中三个是新发现的:GRM5(rs57700751,P = 3.0 × 10 −8)、ALX1(rs765366,P = 4.30 × 10 −8)和DLK1(rs730570,P = 2.53 × 10 −8);还有四个之前报道与 CL/P 相关:IRF6(rs12405750,P = 8.13 × 10 −49)、MYCN(rs4832468,P = 5.21 × 10 −16)、VAX1(rs4752028,P = 3.15 × 10 −8)和MAFB(rs17820943,P = 2.08 × 10 −17)。唇腭裂致病基因鉴定基因解码注意到DLK1和IRF6与 腭裂(CPO)和 唇裂(CLO) 均相关(表 2),因此,共鉴定出13个基因/位点与CPO或唇裂(CLO)相关,其中9个是新基因/位点。
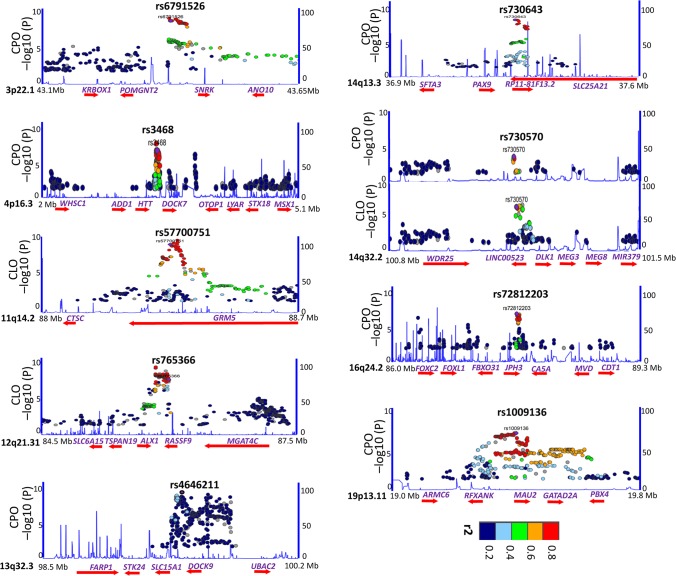
图 2:典型 GWAS 发现的九个新基因座的区域关联
区域关联图表示每个基因座的基因型 SNP 的 −log 10 P值。序列数据与人类 hg19 对齐。Y 轴表示 SNP P值的负对数(以 10 为底),X 轴表示染色体上的位置,底部面板显示 UCSC 基因组浏览器中基因的名称和位置。该区域中具有最低元分析P值的 SNP 以紫色星号标记。其他 SNP 的颜色表示这些 SNP 与领先 SNP 的 r 2。使用 LocusZoom 使用 ASN 人群的 hg19/1000 基因组构建 LD 生成图(2014 年)。
此外,为了评估这些 腭裂(CPO)或 唇裂(CLO) 相关的 13 个基因/位点是否也与 CLP 相关,唇腭裂致病基因鉴定基因解码在一个独立队列中对这 13 个基因/位点的 24 个 SNP 进行了基因分型,该队列由 2,270 例无关的 CLP 散发病例和 3,265 名中国南方汉族对照个体以及 427 名 CLP 患者和 1,832 名中国北方汉族对照个体组成(表 2)。然而,唇腭裂致病基因鉴定基因解码发现只有之前报道的与 CL/P 相关的四个基因(IRF6、MYCN、VAX1和MAFB )与 CLP 显著相关( P < 5 × 10 −8)。这些结果表明,新基因只能通过特定的 非综合征性颌面裂 (NSOFC) 亚型来鉴定以进行关联研究,并且之前鉴定的 CL/P 的遗传因素与 唇裂(CLO) 和 CLP 的遗传因素比与 腭裂(CPO)的遗传因素更接近。
(责任编辑:佳学基因)



