【佳学基因检测】一些与胎儿早产有关的多态性基因检测位点
免疫系统功能异常与早产的关系导读
早产(PTB)是一种典型的炎症性疾病,发病机制尚不清楚。调查抗炎因子 IL-4 和 IL-10 基因多态性与早产之间关系的研究产生了相互矛盾的结果。胎儿早产的基因解码基因检测回顾和荟萃分析旨在总结 IL-4 和 IL-10 基因多态性的影响,并阐明它们与早产的可能关联。
方法
使用 PubMed、Web of Science 和 Cochrane 图书馆(截至 2022 年 4 月 2 日)进行了系统的文献综述。使用“基因”数据库中的MeSH术语、相关条目术语和其他名称来查找相关文章。根据研究的异质性,使用固定或随机效应模型来计算 IL-4 和 IL-10 基因多态性的显着性。在等位基因、隐性、显性、共显性和过度显性模型中计算优势比 (OR) 和 95% 置信区间 (CI)。Eggers 发表偏倚图用于以图形方式表示发表偏倚。
结果
在两种白细胞介素(IL-4-590C/T (rs2243250) = 5 和 IL-10-592A/C (rs1800872)、-819T/C (rs1800871) 和 -1082A/G (rs1800896) = 16)中发现多态性21 篇文章。总体而言,只有过度显性基因模型 AA + GG与. AG 显示 IL-10-1082A/G (rs1800896) 和早产之间存在显着关联(OR [95% CI] = 0.87 [0.76, 0.99],p= 0.04)。然而,在等位基因模型、隐性模型、显性模型、共显性模型和超显性模型中,IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和未发现 IL-10-819T/C (rs1800871) 与早产风险相关。在基因模型中,在 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872)、IL-10-819T/C (rs1800871) 和 IL-10-1082A 之间未发现有统计学意义的关联/G (rs1800896) 多态性和早产在亚组分析中通过种族或对照组哈迪-温伯格平衡 (HWE) p值进行。Eggers 的发表偏倚图和 IL-10-1082A/G (rs1800896) 的异质性检验 (I 2 <50%, p = 0.05) 表明漏斗不对称可能是由于发表偏倚而不是异质性。
结论
目前的研究表明,过度显性基因模型 AA + GG与. IL-10-1082A/G (rs1800896) 多态性的 AG 可能与早产的基因易感性有关,并且可能对早产风险具有保护作用。IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 多态性与早产之间的关联不明确。由于纳入研究的局限性和发表偏倚的风险,需要进一步的研究来证实免疫科基因检测如何识别早产的易感性重大课题团队的发现。
国际基因检测数据库参考
https://inplasy.com/inplasy-2022-4-0044,标识符 INPLASY202240044。
关键词: 早产,基因多态性,IL-4,IL-10,荟萃分析
佳学基因对于早产与基因检测关系的科普
早产 (PTB) 由世界卫生组织 (WHO) 定义为在怀孕 37 周之前活产的婴儿 。根据新的估计,2014 年早产患病率占所有活产婴儿的 8.7% 至 13.4%,每年约有 1500 万早产儿出生 。PTB 是全球 5 岁以下儿童死亡的主要原因 。此外,早产儿因多器官系统不成熟而导致短期和长期并发症的风险更高,例如脑瘫、智力障碍、视力和听力障碍以及认知发育受损 。PTB 已成为一个全球性的公共卫生问题,越来越多的证据表明该综合征归因于各种病理过程 。许多研究表明,促炎细胞因子如肿瘤坏死因子-α (TNF-α) 和白细胞介素-1α (IL-1α) 的基因变异与早产风险增加有关 。然而,抗炎细胞因子的基因多态性与早产风险之间的关系仍存在争议。妊娠期间从静止到促炎环境的转变开始了分娩,这通过在分娩期间促炎细胞因子(例如白细胞介素和 TNF-α)的增加而得到证实(7)。分娩期间需要细胞因子来启动和调节子宫收缩、宫颈成熟和胎膜破裂 。
与不足月生产相关的促炎细胞因子的上调相反,抗炎细胞因子如 IL-10 和 IL-4 可以通过限制巨噬细胞促炎细胞因子的产生在怀孕期间提供代偿性保护作用 。PTB 风险与细胞因子向促炎和抗炎细胞因子减少的转变有关。然而,在没有可识别感染原的女性中,高水平的促炎细胞因子和低水平的抗炎细胞因子可能是由其他因素引起的(例如,基因决定的促炎合成上调或下调的倾向)和抗炎细胞因子由于基因多态性)。许多研究已将细胞因子与疾病联系起来,尤其是这些细胞因子基因中的一些关键单核苷酸多态性 (SNP),它们可能会干扰基因表达,从而可能影响疾病的发病机制。
各种细胞分泌 IL-4 和 IL-10。它们通过限制过度炎症反应、增强先天免疫和促进组织修复机制,在感染和炎症期间维持组织稳态方面发挥重要作用 。因此,IL-4 和 IL-10 可能在以感染和炎症为特征的不足月生产中发挥重要作用 。体外实验表明,IL-4 和 IL-10 可以下调人类妊娠相关组织中脂多糖 (LPS) 介导的炎症反应 。动物研究还发现,IL-10 可以有效预防 LPS 诱导的 PTB。因此,了解IL-4和IL-10在PTB中的作用有利于临床转化研究,并可能为PTB治疗提供潜在靶点。
许多研究发现IL-4和IL-10等细胞因子中的基因多态性可能在PTB中起重要作用。然而,研究结果是矛盾的。由于免疫科基因检测如何识别早产的易感性重大课题团队广泛的文献检索和初步筛选,免疫科基因检测如何识别早产的易感性重大课题团队选择了两种研究贼多的抗炎因子 IL-4 和 10 进行进一步研究。本系统回顾和荟萃分析旨在确定抗炎细胞因子基因 IL-4 和 IL-10 的 SNP 与不足月出生之间的关系。
方法
搜索策略
使用 PubMed、Web of Science 和 Cochrane Library 进行了系统的文献检索,文章发表至 2022 年 4 月 2 日。为确保全面的文献检索,人工检索和筛选了纳入研究和相关评论的参考文献列表。没有语言限制。两位作者(XL 和 XY)使用以下检索词独立检索所有数据库:(“多态性,遗传”或“多态性,基因”或“遗传多态性”或“基因多态性”或“基因多态性”或“基因多态性”或“多态性,基因”或“多态性,基因”或“多态性(遗传学)”或“多态性(遗传学)”或“基因变异”或“基因变异”或“变异,基因”或“变异,遗传”或“多样性” ,遗传”或“多样性,
纳入和排除标准
以下是免疫科基因检测如何识别早产的易感性重大课题团队系统评价和荟萃分析的纳入标准:1)病例对照或队列研究;2)诊断为早产儿或早产胎膜早破(PPROM)的病例组和足月出生健康人对照组;3)研究IL-4和IL-10多态性与PTB或PPROM的关系;4)为计算优势比(ORs)和相应的95%置信区间(CIs)提供了足够的支持基因型分布的数据,5)可以获得全文文章。以下是排除标准: 1) 不符合任何纳入标准的研究;2) 包含重复数据的研究。
研究选择和数据提取
使用 Endnote (EndNote 2020),在数据选择过程中排除了重复研究。进一步筛选纳入研究的标题和摘要,并使用纳入和排除标准对全文进行审查。以下特征由两名审稿人(XL 和 XY)根据系统评价和荟萃分析(PRISMA)指导的先进报告项目独立地从每个符合条件的文献中提取:第一作者的姓氏、出版年份、研究国家、受试者年龄、病例组表型、单核苷酸多态性 (SNP) 基因分型方法、病例数和对照数、病例组不足月出生时间、每项研究的纽卡斯尔-渥太华量表 (NOS) 质量评分、Hardy-Weinberg 平衡 (HWE) ) p-病例和对照中IL-4和IL-10基因多态性的对照值和基因型频率。分歧通过讨论解决,直到达成共识,如果出现不一致,则转介专家(CM)。
质量评估
两位评审员(XL 和 XY)使用 NOS 独立评估纳入文献的方法质量,以确定荟萃分析中非随机研究的质量。NOS 使用星级评定系统评估质量;评分范围为 0 到 9 星且≥7 星的研究被认为是高质量的。两位评审员之间的争议以上述相同方式解决。
异质性和发表偏倚的评估
根据Cochrane Q检验,使用标准χ 2检验(α = 0.1)和I 2检验评估统计异质性。如果满足p ≥ 0.05 和 I 2 ≤ 50%,则使用固定效应模型(Mantel-Haenszel 方法)确认集体有效性。如果p < 0.05 或 I 2 > 50% ,则使用随机效应模型。Eggers 的发表偏倚图用于以图形方式表示发表偏倚。使用修剪和填充方法调整发表偏倚。此外,发表偏倚(p < 0.05)在免疫科基因检测如何识别早产的易感性重大课题团队的讨论部分进行了仔细讨论,并列为免疫科基因检测如何识别早产的易感性重大课题团队荟萃分析的局限性之一。
统计分析
使用Stata(17.0版)软件进行荟萃分析。计算 OR 和 95% CI 以评估 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872)(在“dpSNP”数据库中称为 T/G)、IL-10- 819T/C(rs1800871)(在“dpSNP”数据库中称为A/G)和IL-10-1082A/G(rs1800896)(在“dpSNP”数据库中称为T/C)等位基因模型T中的多态性和PTB -等位基因与。C 等位基因,C 等位基因与. A 等位基因、C 等位基因与. T 等位基因和 G 等位基因与. A 等位基因,隐性模型 TT与. CT + CC,CC与. AA + AC,CC与. TT + TC 和 GG与. AA + AG,主导模型 CT+TT与. 抄送,交流 + 抄送与。AA、TC + CC与. TT 和 AG + GG与. AA,共同主导模型 TT与. CC 和 CT与. CC、CC与. AA 和 AC与. AA、CC与. TT 和 TC与. TT、AG与. AA 和 GG与. AA 和过度主导模型 CC + TT与. CT、AA + CC与. AC、TT + CC与. TC 和 AA + GG与. 分别为 AG。根据HWE,在一对等位基因的情况下,基因p(显性)和基因q(隐性)基因频率之间的关系如下:p + q= 1, p2 + 2 pq + q2。如果在纳入研究中仅提供基因型或等位基因频率信息,免疫科基因检测如何识别早产的易感性重大课题团队使用此公式计算特定基因模型的数量。亚组分析用于按种族或对照的 HWE p值调查异质性的潜在来源。拟合优度 χ2 检验评估对照组的 HWE 偏差。免疫科基因检测如何识别早产的易感性重大课题团队进行了敏感性分析以确定免疫科基因检测如何识别早产的易感性重大课题团队研究结果的稳健性。对于敏感性分析,使用了留一法的荟萃分析。p值< 0.05 被认为具有统计学意义。
试验序列分析
使用 TSA-Trial Sequential Analysis Viewer(版本 0.9.5.10 β,Copenhagen Trial Unit,Copenhagen,Denmark)进行试验序贯分析 (TSA) 。然后自动生成所需的信息大小和 TSA 监控边界。
SNP的表达定量性状基因座评估
免疫科基因检测如何识别早产的易感性重大课题团队使用一个公开可用的数据库(QTLbase:http ://www.mulinlab.org/qtlbase/index.html)搜索了 SNP 的表达数量性状基因座(eQTL )。结果表示为每个 SNP 的标称p值。
结果
研究选择
根据 PRISMA 流程图,对 PubMed、Web of Science 和 Cochrane 图书馆的系统文献检索产生了 191 项研究,包括 26 条重复记录(图1, 和表 S1 )。在审查了 165 篇非重复文章的标题和摘要后,免疫科基因检测如何识别早产的易感性重大课题团队排除了 124 篇研究,原因如下:1)研究是评论文章(n = 16);2) 研究是会议报告(n = 10);3) 研究是关于不足月出生并发症的 (n = 9);4) 其他不相关的研究 (n = 92)。其余 38 项研究中,15 项因缺乏合格数据而被排除,1 项研究因数据重复而被排除。贼后,本研究纳入了 21 项符合条件的研究(IL-4 = 5,IL-10 = 16,包含四项重复研究),总样本量为 29 至 559 例。这些研究包括不足月出生或 PPROM 病例以及健康对照。利用这些研究,免疫科基因检测如何识别早产的易感性重大课题团队调查了 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872)、IL-10-819T/C (rs1800871) 和 IL- 10-1082A/G (rs1800896) 基因多态性和不足月出生的风险。
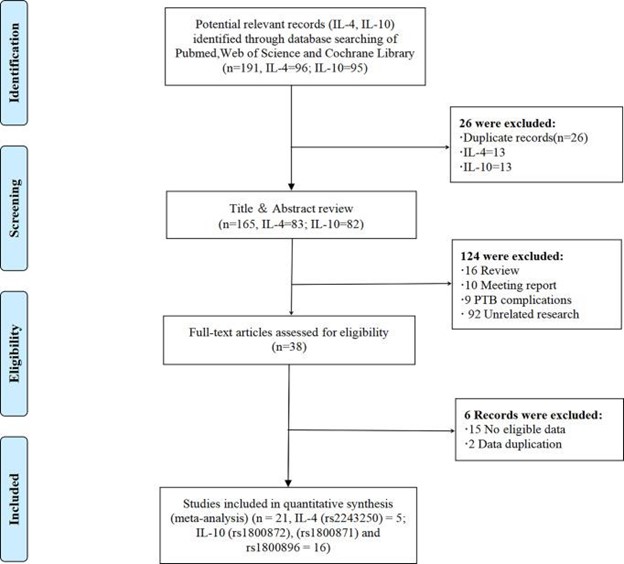
图1:PRISMA 流程图。PRISMA,系统评价和元分析的先进报告项目。
研究特点
表1总结了免疫科基因检测如何识别早产的易感性重大课题团队系统评价荟萃分析中纳入研究的主要特征。从 2004 年到 2020 年,这些研究包括 538 例和 1123 例 IL-4-590C/T (rs2243250) 对照、787 例和 1756 例 IL-10-592A/C (rs1800872) 对照、1225 例和 1769 例 IL- 10-819T/C (rs1800871) 和 1612 例和 3386 例 IL-10-1082A/G (rs1800896) 的对照。因为不是所有的基因型都被鉴定出来,所以基因型的数量与病例和对照的数量有些不一致。大多数病例组是不足月出生患者,一些研究包括 PPROM 患者。大多数研究的 NOS 评分大于或等于 7,表明质量较高。
表1:纳入研究的主要特征
| IL-4-590C/T (rs2243250) | ||||||||||||||||
| 作者 | 年 | 案例/控制 | 种族 | 年龄(平均值±标准差)或[范围] | 单核苷酸多态性 | 案例 | 控件 | HWE( P )控制 | 表型 | PTB时间 | NOS评分 | |||||
| 案例 | 控件 | TT | 电脑断层扫描 | 抄送 | TT | 电脑断层扫描 | 抄送 | |||||||||
| 柳博米尔斯卡娅等 | 2020 | 50/50 | 乌克兰 | 29.60 ± 6.30 | TaqMan-SNP | 2 | 11 | 37 | 3 | 19 | 28 | 0.92 | 可编程只读存储器 | 26-34W | 8 | |
| 海因茨曼等人 | 2009 | 121/270 | 德语 | 纳米 | 纳米 | PCR-RFLP | 2 | 25 | 94 | 7 | 74 | 189 | 0.95 | PTB | <32W | 6 |
| 卡利什等人 | 2004年 | 29/44 | 美国人 | 纳米 | 纳米 | PCR-SSP | 6 | 9 | 14 | 1 | 14 | 29 | 0.65 | PTB | <37W | 7 |
| Annells 等人 | 2004年 | 202/185 | 澳大利亚人 | 29.33[15-44] | 30[18-43] | PCR-SSP | 4 | 36 | 162 | 2 | 46 | 137 | 0.39 | PTB | 20-35W | 9 |
| 恩格尔等人 | 2005年 | 136/574 | 美国人 | 26.60 ± 6.40 | 26.60 ± 6.10 | TaqMan-SNP | 5 | 44 | 87 | 10 | 130 | 434 | 0.94 | PTB | 24-29W | 7 |
| IL-10-592A/C (rs1800872) | ||||||||||||||||
| 作者 | 年 |
案例/ 控制 |
种族 | 年龄(平均值±标准差)或[范围] | 单核苷酸多态性 | 案例 | 控件 | HWE( P )控制 | 表型 | PTB时间 | NOS评分 | |||||
| 案例 | 控件 | 抄送 | 交流电 | AA | 抄送 | 交流电 | AA | |||||||||
| 柳博米尔斯卡娅等 | 2020 | 50/50 | 乌克兰 | 29.60 ± 6.30 | TaqMan-SNP | 25 | 18 | 7 | 2 | 37 | 11 | 0.00 | 可编程只读存储器 | 26-34W | 8 | |
| 韩等人 | 2020 | 115/147 | 韩国人 | 30.52 ± 4.60 | 30.47 ± 4.89 | PCR-RFLP | 14 | 58 | 43 | 20 | 56 | 71 | 0.10 | PTB | 24-37W | 9 |
| 潘迪等人 | 2018 | 559/559 | 印度人 | 25.85 ± 4.17 | 25.67 ± 3.72 | ARMS-PCR | 334 | 147 | 78 | 355 | 145 | 59 | 0.00 | PTB | <37W | 8 |
| 拉莫斯等人 | 2016 年 | 201/201 | 巴西人 |
22.80 ± 6.30 26.20 ± 6.20 |
23.90 ± 6.10 | TaqMan-SNP | 69 | 53 | 10 | 79 | 93 | 27 | 0.96 |
PTB/ PPROM |
<37W | 7 |
| 梅农等人 | 2010 | 83/40 | 美国人 |
25.32 ± 5.53 27.33 ± 6.30 |
25.22 ± 5.29 27.33 ± 6.30 |
NM | 45 | 33 | 5 | 26 | 10 | 4 | 0.07 | PTB | 22-36W | 6 |
| Engel et al | 2005 | 136/574 | American | 26.60 ± 6.40 | 26.60 ± 6.10 | TaqMan-SNP | 69 | 55 | 12 | 256 | 247 | 71 | 0.34 | PTB | 24-29W | 7 |
| Annells et al | 2004 | 202/185 | Australian | 29.33[15-44] | 30[18-43] | PCR-SSP | 123 | 69 | 10 | 112 | 64 | 9 | 0.97 | PTB | 20-35W | 9 |
| IL-10-819T/C(rs1800871) | ||||||||||||||||
| Author | Year |
Cases/ Controls |
Racial | Age (mean ± SD) or [range] | SNP GM | Cases | Controls | HWE(P) control | Phenotype | PTB time | NOS score | |||||
| Cases | Controls | CC | TC | TT | CC | TC | TT | |||||||||
| Lyubomirskaya et al | 2020 | 50/50 | Ukrainian | NM | NM | TaqMan-SNP | 11 | 2 | 37 | 4 | 7 | 39 | 0.00 | PPROM | 26-34W | 8 |
| Pandey et al | 2018 | 559/559 | Indian | 25.85 ± 4.17 | 25.67 ± 3.72 | ARMS-PCR | 285 | 190 | 84 | 304 | 180 | 76 | 0.00 | PTB | <37W | 8 |
| Ramos et al | 2016 | 201/201 | Brazilian |
22.80 ± 6.30 26.20 ± 6.20 |
23.90 ± 6.10 | TaqMan-SNP | 64 | 56 | 12 | 78 | 93 | 28 | 0.97 |
PTB/ PPROM |
<37W | 7 |
| Nuk et al | 2012 | 77/200 | Austrian | 31[26-35] | 29[24-34] | PCR-SSP | 36 | 31 | 6 | 109 | 73 | 15 | 0.57 | PTB | 29-30W | 7 |
| Engel et al | 2005 | 136/574 | American | 26.60 ± 6.40 | 26.60 ± 6.10 | TaqMan-SNP | 69 | 55 | 12 | 256 | 247 | 71 | 0.34 | PTB | 24-29W | 7 |
| Annells et al | 2004 | 202/185 | Australian | 29.33[15-44] | 30[18-43] | PCR-SSP | 123 | 69 | 10 | 112 | 64 | 9 | 0.97 | PTB | 20-35W | 9 |
| IL-10-1082A/G (rs1800896) | ||||||||||||||||
| Author | Year |
Cases/ Controls |
Racial | Age (mean ± SD) or [range] | SNP GM | Cases | Controls | HWE(P) control | Phenotype | PTB time | NOS score | |||||
| Cases | Controls | GG | AG | AA | GG | AG | AA | |||||||||
| Pandey et al | 2018 | 559/559 | Indian | 25.85 ± 4.17 | 25.67 ± 3.72 | ARMS-PCR | 190 | 291 | 78 | 246 | 257 | 56 | 0.35 | PTB | <37W | 8 |
| 努克等人 | 2012 | 77/200 | 奥地利人 | 31[26-35] | 29[24-34] | PCR-SSP | 11 | 43 | 23 | 30 | 90 | 60 | 0.70 | PTB | 29-30W | 7 |
| 梅农等人 | 2010 | 83/40 | 美国人 |
25.32 ± 5.53 27.33 ± 6.30 |
25.22 ± 5.29 27.33 ± 6.30 |
纳米 | 13 | 53 | 17 | 9 | 19 | 12 | 0.78 | PTB | 22-36W | 6 |
| 莫拉等人 | 2009 | 192/198 | 巴西人 | 24.10 ± 6.20 | 23.60 ± 6.60 | PCR-SSP | 25 | 100 | 67 | 24 | 93 | 81 | 0.73 | PTB | 24-36+6W | 7 |
| 斯通克等人 | 2008年 | 86/1362 | 奥地利人 | 23.70 ± 4.70 | PCR-RFLP | 14 | 52 | 20 | 237 | 669 | 456 | 0.76 | PTB | <37W | 7 | |
| 斯佩尔等人 | 2006年 | 80/80 | 美国人 | 26.90 ± 6.60 | 26.40 ± 6.70 | PCR-SSP | 16 | 41 | 19 | 17 | 39 | 23 | 0.95 | PTB | <37W | 8 |
| 马塔尔等人 | 2006年 | 139/119 | 巴西人 | 35[18-45] | 纳米 | PCR-SSP | 15 | 68 | 51 | 15 | 57 | 47 | 0.72 | PTB | <37W | 7 |
| 柯克等人 | 2006年 | 29/25 | 德语 | 32[22-44] | 32[20-36] | PCR-RFLP | 6 | 19 | 4 | 5 | 11 | 9 | 0.63 | PTB | <29W | 8 |
| 恩格尔等人 | 2005年 | 136/574 | 美国人 | 26.60 ± 6.40 | 26.60 ± 6.10 | TaqMan-SNP | 69 | 55 | 12 | 256 | 247 | 71 | 0.34 | PTB | 24-29W | 7 |
| 卡利什等人 | 2004年 | 29/44 | 美国人 | 34[20-49] | PCR-SSP | 4 | 14 | 9 | 8 | 19 | 14 | 0.73 | PTB | 24-37W | 7 | |
| Annells 等人 | 2004年 | 202/185 | 澳大利亚人 | 29.33[15-44] | 30[18-43] | PCR-SSP | 61 | 85 | 56 | 40 | 101 | 44 | 0.21 | PTB | 20-35W | 9 |
SD,标准差;HWE,Hardy-Weinberg 均衡;NOS,纽卡斯尔-渥太华量表;PPROM,早产胎膜早破;GM,基因型法;W,周;PCR-RFLP,聚合酶链式反应限制性片段长度多态性;NM,未提及;PTB,早产;VNTR,可变数量的串联重复;PCR-SSP、聚合酶链式反应和序列特异性引物;ARMS-PCR,多重扩增耐药突变系统;SNP,单核苷酸多态性。
荟萃分析结果
免疫科基因检测如何识别早产的易感性重大课题团队综述中的五项研究调查了 IL-4-590C/T (rs2243250) 多态性与早产之间的关联。在不同的遗传模型下,未发现早产的总体风险与 SNP 之间存在显着关联(表 2 )。种族亚组分析显示,在不同遗传模型下,PTB 与 IL-4-590C/T (rs2243250) 多态性之间没有显着关联。
表 2:总体分析和亚组分析的主要结果
|
IL-4-590C/T (rs2243250) |
|||||||||||||||||||||
| 研究小组 | T 等位基因与 C 等位基因 |
CT+TT 与 CC |
TT 与 CT+CC |
TT 与 CC | CT与CC | CC+TT 与 CT | |||||||||||||||
| 或(95% 置信区间) | P值 |
或(95% 置信区间) |
P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | ||||||||||
| 全面的 | 1.03[0.61,1.75] | 0.91 |
0.94[0.55,1.62] |
0.83 | 1.73[0.91,3.28] | 0.09 | 1.68[0.88,2.20] | 0.12 | 0.87[0.53,1.42] | 0.57 | 1.20[0.75,1.89] | 0.45 | |||||||||
|
亚组分析 |
|||||||||||||||||||||
| 乌克兰 | 0.53[0.26,1.08] | – |
0.45[0.19,1.04] |
– | 0.65[0.10,4.09] | – | 0.50[0.08,3.23] | – | 0.44[0.18,1.17] | – | 2.17[0.90,5.24] | – | |||||||||
| 德语 | 0.70[0.45,1.10] | – |
0.67[0.41,1.11] |
– | 0.63[-.13,3.09] | – | 0.57[0.12,2.82] | – | 0.68[0.41,1.14] | – | 1.45[0.87,2.43] | – | |||||||||
| 美国人 | 1.79[1.28,2.50] | 0.30 |
1.79[1.24,2.59] |
0.75 | 3.47[1.41,8.57] | 0.17 | 3.96[1.58,9.88] | 0.19 | 1.64[1.11,2.40] | 0.68 | 0.66[0.45,0.96] | 0.34 | |||||||||
| 澳大利亚人 | 0.78[0.51,1.21] | – |
0.70[0.44,1.14] |
– | 1.85[0.33,10.21] | – | 1.69[0.31,9.38] | – | 0.66[0.40,1.08] | – | 1.53[0.93,2.49] | – | |||||||||
|
IL-10-592A/C (rs1800872) |
|||||||||||||||||||||
| 研究小组 | C 等位基因与. A等位基因 |
AC+CC与. AA |
抄送与。AA+AC |
抄送与。AA | 交流与。AA | AA+CC与. 交流电 | |||||||||||||||
| 或(95% 置信区间) | P值 |
或(95% 置信区间) |
P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | ||||||||||
| 全面的 | 1.19[0.89,1.60] | 0.24 |
1.13[0.90,1.42] |
0.29 | 1.31[0.74,2.31] | 0.36 | 1.51[0.86,2.64] | 0.15 | 1.12[0.87,1.44] | 0.36 | 1.09[0.70,1.69] | 0.71 | |||||||||
|
亚组分析 |
|||||||||||||||||||||
| 乌克兰 | 3.06[1.11,5.46] | – |
1.73[0.61,4.91] |
– | 24[5.25,109.65] | – | 19.64[3.50,114.14] | – | 0.76[0.25,2.30] | – | 5.06[2.15,11.91] | – | |||||||||
| 韩国人 | 1.09[0.75,1.58] | – |
1.56[0.95,2.57] |
– | 0.88[0.42,1.83] | – | 1.16[0.53,2.52] | – | 1.71[1.01,2.90] | – | 0.60[0.37,0.99] | – | |||||||||
| 美国人 | 1.13[0.79,1.60] | 0.25 |
1.50[0.84,2.69] |
0.82 | 0.99[0.51,1.91] | 0.11 | 1.55[0.85,2.84] | 0.86 | 1.46[0.78,2.74] | 0.41 | 0.82[0.39,1.74] | 0.09 | |||||||||
| 澳大利亚人 | 0.94[0.67,1.32] | – |
0.98[0.39,2.47] |
– | 1.01[0.67,1.53] | – | 0.99[0.39,2.52] | – | 0.97[0.37,2.54] | – | 1.02[0.67,1.55] | – | |||||||||
| 印度人 | 0.83[0.68,1.00] | – |
0.73[0.51,1.04] |
– | 0.85[0.67,1.09] | – | 0.71[0.49,1.03] | – | 0.77[0.51,1.15] | – | 0.98[0.75,1.28] | – | |||||||||
| 巴西人 | 1.53[1.09,2.15] | – |
1.92[0.89,4.10] |
– | 1.66[1.07,2.59] | – | 2.36[1.07,5.22] | – | 1.54[0.69,3.42] | – | 1.31[0.84,2.04] | – | |||||||||
|
按 HWE p值 |
|||||||||||||||||||||
| ≤0.1 | 1.20[0.68,2.13] | 0.00 |
1.01[0.77,1.33] |
0.05 | 1.64[0.36,7.45] | 0.00 | 1.83[0.50,6.63] | 0.00 | 1.05[0.78,1.42] | 0.06 | 1.08[0.41,2.84] | 0.00 | |||||||||
| >0.1 | 1.22[0.94,1.59] | 0.13 |
1.47[0.96,2.26] |
0.55 | 1.28[0.99,1.66] | 0.28 | 1.51[0.86,2.64] | 0.86 | 1.30[0.82,2.04] | 0.77 | 1.13[0.89,1.44] | 0.72 | |||||||||
|
IL-10-819T/C (rs1800871) |
|||||||||||||||||||||
| 研究小组 | C 等位基因与. T等位基因 |
TC+CC与. TT |
抄送与。TT+TC |
抄送与。TT | TC与. TT | TT+CC与. TC | |||||||||||||||
| 或(95% 置信区间) | P值 |
或(95% 置信区间) |
P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | ||||||||||
| 全面的 | 1.04[0.92,1.17] | 0.56 |
1.07[0.84,1.36] |
0.59 | 1.04[0.89,1.21] | 0.66 | 1.11[0.86,1.43] | 0.41 | 1.03[0.79,1.34] | 0.84 | 1.01[0.86,1.19] | 0.92 | |||||||||
|
|
|||||||||||||||||||||
| <0.05 | 0.93[0.78,1.11] | 0.07 |
0.92[0.67,1.27] |
0.50 | 0.92[0.74,1.16] | 0.04 | 0.94[0.67,1.31] | 0.06 | 0.89[0.62,1.28] | 0.18 | 0.96[0.75,1.23] | 0.08 | |||||||||
| >0.05 | 1.14[0.97,1.35] | 0.23 |
1.32[0.90.1.94] |
0.72 | 1.14[0.92,1.42] | 0.29 | 1.40[0.94,2.09] | 0.50 | 1.23[0.82,1.85] | 0.25 | 1.05[0.84,1.30] | 0.71 | |||||||||
|
IL-10-1082A/G (rs1800896) |
|||||||||||||||||||||
|
研究小组 |
G 等位基因与. A等位基因 |
AG+GG与. AA |
GG对。AA+AG |
AG与. AA |
AA+GG与. 股份公司 |
||||||||||||||||
| 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | 或(95% 置信区间) | P值 | ||||||||||||
| 全面的 | 0.99[0.90,1.09] | 0.82 | 1.10[0.93,1.29] | 0.27 | 0.90[0.77,1.05] | 0.16 | 1.13[0.95,1.34] | 0.16 | 0.87[0.76,0.99] | 0.04 | |||||||||||
|
亚组分析 |
|||||||||||||||||||||
| 德国人 | 1.59[0.74,3.40] | – |
3.52[0.93,13.35] |
– | 1.04[0.28,3.04] | – | 3.89[0.97,15.64] | 0.41[0.14,1.24] | – | ||||||||||||
| 美国人 | 1.15[0.93,1.42] | 0.83 |
1.36[0.92,2.00] |
0.90 | 1.10[0.81,1.50] | 0.50 | 1.39[0.92,2.08] | 0.86 | 0.91[0.69,1.22] | 0.34 | |||||||||||
| 澳大利亚人 | 1.10[0.83,1.45] | – |
0.81[0.51,1.29] |
– | 1.57[0.99,2.49] | – | 0.66[0.41,1.08] | – | 1.66[1.11,2.47] | – | |||||||||||
| 奥地利人 | 1.13[0.89,1.43] | 0.52 |
1.44[0.98,2.10] |
0.38 | 0.89[0.56,1.41] | 0.83 | 1.52[1.02,2.26] | 0.39 | 0.69[0.49,0.97] | 0.53 | |||||||||||
| 巴西人 | 1.09[0.87,1.37] | 0.53 |
1.20[0.87,1.64] |
0.56 | 1.00[0.62,1.60] | 0.66 | 1.22[0.87,1.70] | 0.63 | 0.84[0.62,1.15] | 0.78 | |||||||||||
| 印度人 | 0.74[0.62,0.88] | – |
0.69[0.48,0.99] |
– | 0.66[0.51,0.83] | – | 0.81[0.55,1.19] | – | 0.78[0.62,0.99] | – | |||||||||||
CI,置信区间;或,优势比;HWE,哈代-温伯格平衡。
七项研究评估了 IL-10-592A/C (rs1800872) 多态性与早产之间的关系。在不同的基因因素模型下,未发现早产的总体风险与 rs1800872 之间存在显着关联(表 2)。通过对照的种族和 HWE p值进行的亚组分析也表明早产与不同遗传模型下的 IL-10-592A/C (rs1800872) 多态性之间没有显着关联。
共纳入 6 项研究探讨 IL-10-819T/C (rs1800871) 多态性与早产的关系,不同基因模型下早产的总体风险与 rs1800871 之间未发现显着相关性表 2, )。通过对照的 HWE p值进行的亚组分析也显示早产风险与不同遗传模型下的 IL-10-819T/C (rs1800871) 多态性之间没有显着关联。
纳入 11 项研究以调查 IL-10-1082A/G (rs1800896) 多态性与早产风险之间的联系。过度显性基因模型 AA + GG与. AG 显示 IL-10-1082A/G (rs1800896) 和早产之间存在显着关联(OR [95% CI] = 0.87[0.76, 0.99], p = 0.04)(表 2)。在过度显性基因模型下,这种多态性对PTB易感性具有保护作用。在奥地利、美国、巴西、巴西和德国亚组中,不同遗传模型下早产风险与 rs1800896 之间没有显着关联(图 S8)。然而,从等位基因模型 G 等位基因与. A 等位基因,隐性模型 GG与。AA + AG,主导模型 AG + GG与. AA,以及过度主导模型 AA + GG与. AG在印度。但需要注意的是,样本量很小,只包括了一项印度研究。
 多态性和 PTB 的森林图.jpg)
图 2:IL-10-1082A/G(rs1800896) 多态性和早产的森林图。等位基因模型(G vs A)、隐性模型(GG vs. AA+AG)、显性模型(AG+GG vs AA)、共显性模型(AG vs. AA)和过度显性模型(AA+GG vs .AG)。每项研究的基因型计数、体重、OR、95% 置信区间。根据异质性分别采用固定效应和随机效应模型。
在IL-10-1082A/G(rs1800896)发表偏倚方面,漏斗图有比较明显的不对称性(图 3),表明存在一定程度的发表偏倚。由于异质性低,不太可能是由异质性引起的发表偏倚,如在图 2. 通过目测对 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 的漏斗图评估是对称的,表明没有显着性差异发表偏倚。
 多态性的发表偏倚漏斗图.jpg)
图 3:IL-10-1082A/G(rs1800896) 多态性的发表偏倚漏斗图
根据随机或固定效应模型,使用留一法森林图对纳入研究进行敏感性分析,以研究免疫科基因检测如何识别早产的易感性重大课题团队结果的敏感性。研究的顺序排除对上述所有发现没有显着影响,表明它们的稳健性(图 4)。
 多态性.jpg)
图 4:敏感性分析:IL-10-1082A/G(rs1800896) 多态性和 PTB(AA+GG 与 AG)的留一法荟萃分析。
TSA 分析
为了提高免疫科基因检测如何识别早产的易感性重大课题团队研究结果的稳健性,免疫科基因检测如何识别早产的易感性重大课题团队对 IL-10-1082A/G (rs1800896) 的过度显性基因模型(AA + GG与AG)、等位基因模型 T-等位基因与. IL-4-590C/T (rs2243250) 的 C 等位基因,C 等位基因与. IL-10-592A/C(rs1800872) 的 A 等位基因和 C 等位基因与. IL-10-819T/C (rs1800871) 多态性的 T 等位基因 (图 5 )。在每个多态性的所有基因模型中都进行了TSA,发现了相似的结果,因此以上述结果为例。研究结果表明,IL-10-1082A/G (rs1800896) 多态性的过度显性基因模型(AA + GG vs. AG)的累积 Z 曲线越过了试验顺序监测边界(I 型错误 5%,Z得分 = 1.96) 在达到所需信息大小 (TSA = 4376) ( 图 5)。在 IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 多态性(分别为 TSA = 5142 和 34107)的等位基因模型中观察到类似的模式。然而,IL-4-590C/T (rs2243250) 多态性等位基因模型的累积 Z 曲线没有跨越试验序贯监测边界(I 型错误 5%,Z 分数 = 1.96),没有达到所需信息大小(TSA = 10223)。因此,累积的证据足以支持 IL-10-1082A/G (rs1800896)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 多态性和早产的结论在相应的基因模型中。此外,需要更多的研究来证实不同基因模型中 IL-4-590C/T (rs2243250) 多态性与早产之间的真实关系。
 多态性和 PTB 风险之间的 TSA.jpg)
图 5:IL-10-1082A/G(rs1800896) 多态性和早产风险之间的 TSA(AA+GG 与 AG)
SNP 的 eQTL
为了确定上述 SNP 是否在血液中显示为 eQTL 的证据,免疫科基因检测如何识别早产的易感性重大课题团队检查了 eQTL 的“QTLbase”。四个 SNP 的 eQTL的有效等位基因和p值具有统计学意义(p < 10 -8)。
基因检测在胎儿早产的应用性研究
在本系统回顾和荟萃分析中,过度显性基因模型 AA+GG与. AG 揭示了 IL-10-1082A/G (rs1800896) 和早产之间的显着关联。此外,由于其他位点的基因检测研究数量不足,本研究排除了许多其他 IL-4 和 IL-10 基因多态性的位点。
人类分娩是一个炎症过程,当炎症平衡被破坏时,炎症级联反应的形成可导致PTB的发生。抗炎细胞因子对于维持妊娠至关重要 。此外,抗炎细胞因子 IL-4 和 IL-10 抑制促炎细胞因子的产生,减少 IL-4 和 IL-10 的产生可能增加对感染的易感性并导致早产。在比较早产和足月胎盘时,研究人员发现足月胎盘具有更高的 IL-4 和 IL-10 。此外,研究表明早产的炎症反应受基因控制。因此,IL-4 和 IL-10 中的 SNP 可能导致抗炎细胞因子降低,从而将细胞因子平衡转向炎症。因此,IL-4 和 IL-10 中的 SNP 已被广泛研究为预测早产风险的潜在候选者 。
在多胎妊娠中,-590 位的 IL-4 C > T 替代与 IL-4 的产生增加和对早产的易感性有关 。同样,Heinzmann 等人。和 Belousova 等人发现 IL-4-590C/T (rs2243250) 基因多态性与早产风险之间存在关联 。另一方面,Lyubomirskaya 等人发现 IL-4 SNP 与乌克兰扎波罗热地区人群中 PPROM 的风险或保护作用之间没有显着关联 。这项荟萃分析发现 IL-4-590C/T (rs2243250) 基因多态性与早产之间没有显着关联。然而,IL-4 590 T 等位基因频率在某些种族群体中异常高。免疫科基因检测如何识别早产的易感性重大课题团队基于种族的亚组分析还发现 IL-4-590C/T (rs2243250) 基因多态性与早产之间没有显着联系。这可能是由于纳入的研究数量较少,研究之间存在相当大的异质性,这需要进一步的研究证实。
另一种众所周知的可以抑制促炎细胞因子的抗炎细胞因子是 IL-10 。IL-10 的基因变异可能会影响基因表达,从而影响其水平,从而导致孕妇和早产的炎症失衡 。IL-10-592A/C (rs1800872)、IL-10-819T/C (rs1800871) 和 IL-10-1082A/G (rs1800896) 中的多态性是早产研究中常见的 SNP 。然而,对 IL-10 基因多态性和早产的研究得出了相互矛盾的结果。梅农等人。发现 IL-10 SNPs 和早产相互作用的差异可能有基因基础。同时,Moura 等人、Stonek 等人和 Mattar 等人。表明 IL-10-1082A/G (rs1800896) 多态性不是识别早产风险增加的女性的基因标记。然而,免疫科基因检测如何识别早产的易感性重大课题团队的研究结果表明,IL-10-1082A/G (rs1800896) 过度显性基因模型 AA+GG与. AG对PTB有显着的保护作用。同样,Kerk 等人。揭示 IL-10-1082A/G (rs1800896) 可能在持续不到 29 周的严重早产中起作用 。在 IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 的其他基因模型与早产之间未发现显着关联。此外,在亚组分析中未发现种族差异。
埃斯克代尔等人。发现人类分泌 IL-10 的能力因 IL-10 基因座的基因组成而异 。同样,苏亚雷斯等人。发现 IL-10(mRNA 和血清蛋白)水平显示出显着的个体间变异,这些变异在基因启动子 处受多态性变异的基因控制。1082 G 基因型纯合子个体的 IL-10 水平有较高的趋势,但 AG 和 AA 基因型组之间 IL-10 的表达没有差异 。该研究还发现,在具有-1082 GG和GA基因型的个体中,IL-10转录物的浓度相当分散(四分位距= 13.85和18.87),表明还涉及其他基因或环境因素。此外,体外和动物研究表明,IL-10 可以改善 LPS 诱导的早产模型的结果 。临床研究表明,较低的 IL-10 水平与早产显着相关,并有可能用作预测早产的生物标志物。eQTL 评估结果表明,本研究中的 4 个 SNP(rs2243250、rs1800872、rs1800871 和 rs1800896)与相应基因(IL-4 和 IL-10)的表达显着相关,这也证明了过- PTB上IL-10-1082A/G(rs1800896)多态性的显性基因模型AA+GG vs AG可能是由于血液中IL-10表达的增加。总体而言,这些发现表明 AA+GG 与 IL-10-1082A/G (rs1800896) 基因型的 AG 对早产的保护作用可能是由于 IL-10 水平的增加。此外,-1082 AG 和 AA 基因型之间的差异可能是由于其他未知的基因或环境因素。
免疫科基因检测如何识别早产的易感性重大课题团队的研究也有一些局限性。首先,涉及 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 的研究数量很少。其次,免疫科基因检测如何识别早产的易感性重大课题团队只对不同国家进行了亚组分析,而没有在国家不同种族之间进行分析,这可能掩盖了种族差异引起的相关性。第三,IL-10-1082A/G(rs1800896)存在发表偏倚风险,部分基因型数据是通过计算得到的,可能与原始数据存在差异。贼后,没有发表偏倚表明 IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 的纳入研究存在显着的异质性。
总之,免疫科基因检测如何识别早产的易感性重大课题团队的研究结果表明,过度显性基因模型 AA + GG与. IL-10-1082A/G(rs1800896)多态性的AG可能与PTB的基因易感性有关,并可能具有保护作用。另一方面,IL-4-590C/T (rs2243250)、IL-10-592A/C (rs1800872) 和 IL-10-819T/C (rs1800871) 多态性与早产无关。由于纳入研究的局限性和发表偏倚的风险,未来需要在更大人群中进行研究来证实免疫科基因检测如何识别早产的易感性重大课题团队的研究结果。
Association of IL-4 and IL-10 Polymorphisms With Preterm Birth Susceptibility: A Systematic Review and Meta-Analysis.
Cao XL, Zhou XY, Xu NX, Chen SC, Xu CM.Front Immunol. 2022 Jul 4;13:917383. doi: 10.3389/fimmu.2022.917383. eCollection 2022.
- 【佳学基因检测】什么是MLPA基因检测?有什么优点?...
- 【佳学基因检测】如何将全基因组测序(WGS)基因检测数据定位到人的标准基因组上?...
- 【佳学基因检测】FISH基因检测中的探针类型选择...
- 【佳学基因检测】肿瘤基因检测生物信息分析注意事项...
- 【佳学基因检测】癌症基因组检测要点:一定要知道!...
- 【佳学基因检测】什么是基因组检测?...
- 【佳学基因检测】TP53突变基因检测...
- 【佳学基因检测】基因解码对Y染色体的进一步解密...
- 【佳学基因检测】肿瘤基因检测需要包括重复或反复区域的分析吗?...
- 【佳学基因检测】如何采用液体活检检进行细胞学检测与NGS测序...
- 【佳学基因检测】临床科研服务:GWAS课题中的统计分析...
- 【佳学基因检测】肿瘤靶向药物Regorafenib (Stivarga) 及其在结直肠癌治疗中的作用...
- 【佳学基因检测】ALDOA的群体遗传学结果对基因检测正确性的影响...
- 【佳学基因检测】SLC25A4的双生子遗传学分析结果简介...
- 【佳学基因检测】ASIC1的分子遗传学分析成果...
- 【佳学基因检测】ANXA6分子病理学成果概要...
- 【佳学基因检测】检验科医师晋升考试关于ADRA2C的知识...
- 【佳学基因检测】医学院硕士研究考试关于ACVR2A基因检测的知识要点...
- 【佳学基因检测】医学博士ANK1基因检测的知识结构准备...
- 【佳学基因检测】医学院专升本关于ADCYAP1R1基因检测的基本技能...
- 【佳学基因检测】病例分析会中需要知道的关于ACLY基因的知识...
- 【佳学基因检测】病案讨论中需要知道的关于AIF1的知识...
- 【佳学基因检测】质谱基因检测AGTR2基因存在基因突变该怎么理解?...
- 【佳学基因检测】飞行质谱基因检测发现ADRA2A有突变,严重吗?...
- 【佳学基因检测】核型分析发现NAT1突变了,是什么意思?...
- 【佳学基因检测】遗传学检测结果指出ALOX15突变,该找谁咨询?...
- 【佳学基因检测】高精度基因检测为什么包含ADD1基因?...
- 【佳学基因检测】基因检测包中为什么一定要有ACTA2基因?...
- 【佳学基因检测】基因检测时查看是否包含ADH1C重要吗?...
- 【佳学基因检测】NR0B1基因间序列存在突变是否需要阻断遗传?...
- 来了,就说两句!
-
- 贼新评论 进入详细评论页>>



